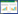文章信息
- 林志群,赵大川,陈飞,黄宗海,杨少华,赵海军,莫林海.
- LIN Zhiqun, ZHAO Dachuan, CHEN Fei, HUANG Zonghai, YANG Shaohua, ZHAO Haijun, MO Linha.
- AT模体结合因子1对结直肠癌LOVO细胞增殖侵袭的影响
- Effect of AT Motif Binding Factor 1 on Proliferation and Invasion of Colorectal Carcinoma Cell Line LOVO
- 肿瘤防治研究, 2016, 43(4): 267-271
- Cancer Research on Prevention and Treatment, 2016, 43(4): 267-271
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2016.04.006
-
文章历史
- 收稿日期: 2015-09-22
- 修回日期: 2015-11-30
结直肠肿瘤的发病率呈逐年上升状态。AT模体结合因子1(AT motif binding factor 1,ATBF1)基因,是近年来新发现的抑癌基因[1]。该基因定位于人16q22.3-q23.1,其表达产物能和甲胎蛋白(Alpha fetal protein,AFP)基因的启动子和增强子AT富集区结合,调节AFP的表达[2]。在ATBF1转录时,由于其mRNA选择性剪接,可产生2种异构体,即ATBF1-A和ATBF1-B,ATBF1-A是ATBF1基因发挥生物学效应的主要产物,相对分子质量高达404 kD,是目前相对分子质量最大的反式作用因子,它能通过结合AFP的启动子,抑制其增强子,从而抑制AFP的表达,并诱导细胞分化与凋亡[3]。
目前研究发现,ATBF1在神经系统、乳腺癌、胃癌、肝癌、前列腺癌、头颈鳞状细胞癌、皮肤癌[1, 4, 5, 6, 7, 8]等组织中呈现抑癌基因特征,通过不同机制影响肿瘤的发生、发展。在我们前期对结直肠癌临床病理样本的研究发现,结直肠癌组织与癌旁组织的ATBF1蛋白及mRNA水平上存在差异化的表达[9]。本研究采用瞬时转染法构建高表达ATBF1的人结直肠癌细胞(LOVO)模型,并利用此模型在体外水平探讨了ATBF1在调控人结直肠癌细胞增殖、凋亡及侵袭性的作用,并初步阐明其调控机制。
1 材料和方法 1.1 主要材料RPMI1640购自美国Hyclone公司。胎牛血清(fetal bovine serum,FBS)购自杭州四季青公司。脂质体Lipofectamine2000购自美国Gibco公司。TRIzol、CCK-8购自日本TaKaRa公司。RT-PCR试剂盒购自北京天根生化科技有限公司。ATBF1、Notch3兔抗人单克隆抗体均购自英国Abcom公司。真核表达载体pEGFP-ATBF1,空白载体pEGFP均购自美国Clontech公司。结直肠癌细胞LOVO由南方医科大学细胞生物学教研室保存。
1.2 方法 1.2.1 细胞培养LOVO细胞在37℃、5%CO2饱和湿度的细胞培养箱中培养,采用含有10%小牛血清的RPMI1640培养液。
1.2.2 质粒转染实验实验组采用脂质体Lipofectamine2000,将质粒pEGFP-ATBF1转染入人结肠癌LOVO细胞,对照组为Lipofectmine2000将空白质粒pEGFP转染入LOVO细胞,空白组只采用Lipofectmine2000作用,具体转染操作按试剂盒说明书进行。转染24~72 h后观察报告基因绿色荧光蛋白在细胞中的表达。
1.2.3 实时定量荧光PCR检测各组细胞ATBF1的转染情况将pEGFP-ATBF1转染LOVO细胞,并设空白组和对照组,用TRIzol提取总RNA,测定样本A260/A280及样本浓度。通过北京天根生化科技有限公司的 FastQuant RT Kit(with gDNase)试剂盒进行反转录合成cDNA。由上海生工生物工程有限公司设计并合成ATBF1、内参ACTB引物:ATBF1上游引物: 5’-CAAGTTCACGACGGACAACCT-3’;下游引物:5’-GCTTGCACTGGTATGAGTCCC-3’。ACTB上游引物:5’-GAA ATC GTG CGT GAC ATT AAG-3’; 下游引物: 5’-CTA GAA GCA TTT GCG GTG GAC GAT GGA GGG GCC-3’。按照天根公司SuperReal PreMix Plus试剂盒说明书,通过Bio-Rad公司PCR仪器(7500)SYBR ® Green分析法进行荧光定量PCR检测。反应条件:(1) 95℃ 15 min;(2) 95℃ 10 s,60℃ 60 s,40个循环;(3) 95℃ 15 s,55℃ 45 s,95℃ 15 s。最后以2-ΔΔCT法计算基因相对表达量。
1.2.4 Western blot检测分析采用蛋白印迹法,按照凯基全蛋白提取试剂盒(KGP2100)说明书提取转染后的各组细胞全蛋白。按照BCA蛋白浓度检测试剂盒(南京凯基,KGPBCA)说明书对所提取的总蛋白进行定量。蛋白产物经离心、蛋白变性后,加入Loading buffer上样至SDS-PAGE胶电泳分离,PVDF膜恒流电转,5%脱脂奶粉封闭1 h,Tween-PBS洗膜后加入一抗,4℃过夜孵育,再次洗膜后加入二抗,室温孵育1 h,Tween-PBS洗膜,ECL化学发光法显影,将显影得到的条带进行灰度分析,并通过相对灰度值统计ATBF1、Notch3的表达情况。实验选用GAPDH为内参对照。
1.2.5 CCK-8测定细胞的生长曲线将LOVO细胞接种于96孔板,每孔约5 000个细胞,培养贴壁6 h后,按说明书将pEGFP-ATBF1转染入LOVO,设立对照组和空白组,观察转染24、48、72 h后细胞生长情况。在上述各时间点终止培养,每孔加入10 μl CCK-8及100 μl基础培养液,37℃、5%CO2继续培养1 h,置酶标仪上测OD450值,每组5个平行孔。
1.2.6 流式细胞术测定细胞凋亡pEGFP-ATBF1转染LOVO,并设空白组和对照组,48 h后收集约2×106个细胞,l 000 r/min离心10 min,PBS冲洗,加入70%冷乙醇1.5 ml,4℃固定过夜,流式细胞仪检测细胞凋亡情况。
1.2.7 体外侵袭实验Transwell板上下室之间隔以直径为3 mm无聚乙烯吡咯烷酮的聚碳酸酯膜(孔径为8 μm),膜上铺匀1:2稀释的基膜胶(Matrige1)。下室加入含10%胎牛血清的完全培养液600 μl。上室加入200 μl各组细胞悬液。置于37℃、5%CO2培养箱中培养12 h,弃去上室液体,取出聚碳酸酯膜,用棉签仔细擦尽膜上Matrigel胶及未穿膜的细胞,甲醛固定5 min,0.1%结晶紫染色后,400倍光学显微镜下计数穿过聚碳酸酯膜的细胞数。随机计数5个视野内的细胞数,取平均值进行统计处理。迁移率=穿过膜的细胞数/总细胞数×100%。
1.3 统计学方法应用GeneTools对RT-PCR和Western blot结果进行灰度值分析,上述实验均分别重复三次。应用SPSS21.0统计软件进行统计学处理,统计学数据用(x±s)表示,两样本均数比较采用t检验,多个样本均数比较应用单因素方差分析(One-way ANOVA),P<0.05为差异有统计学意义。
2 结果 2.1 细胞的显微镜下改变未经处理的LOVO细胞完整,大小均匀,贴壁后呈长梭形。经pEGFP-ATBF1转染48 h后LOVO细胞形态发生改变,一部分细胞体积变小,突出减少,呈椭圆形,细胞之间稍有脱离。而对照组,即pEGFP转染的LOVO细胞无明显改变。免疫荧光结果显示,转染入细胞的pEGFP-ATBF1在24 h已在LOVO内表达,见图 1。

|
| 图 1 转染24h后的LOVO细胞 Fig. 1 Status of LOVO cells 24h after transfection |
运用2-△△CT计算出各组细胞的ATBF1 mRNA水平与空白组的相对表达量,结果显示,实验组的ATBF1 mRNA量明显高于空白组与对照组(P<0.001),而空白组与对照组的ATBF1 mRNA差异无统计学意义。与此相反实验组的Notch3 mRNA低于空白组与对照组(P<0.001)。
|
| 图 2 ATBF1和Notch3 mRNA的RT-PCR分析结果 Fig. 2 RT-PCR analysis of ATBF1 and Notch3 mRNA |
与PCR检测结果一致,Western blot结果同样可以看出,实验组ATBF1蛋白表达量明显高于空白组与对照组(P<0.001)。而空白组与对照组的ATBF1差异无统计学意义。实验组Notch3的表达量低于空白组与对照组(P<0.05),见图 3。

|
| 图 3 ATBF1、Notch3的Western blot分析结果 Fig. 3 Western blot analysis of ATBF1 and Notch3protein |
在转染后24 h内,三组细胞均有不同的增殖趋势,但实验组组并没有明显的增殖趋势,24 h时OD450值明显低于其他两组。48 h后,空白组与对照组OD450值仍在持续增长,表示细胞数也在增长,而实验组OD450值增长缓慢,表示细胞数增加不明显,提示细胞生长受到抑制,甚至可能存在细胞的凋亡、坏死。72 h后,空白组与对照组OD450值达到峰值,而实验组OD450值已无明显增长趋势,见图 4。
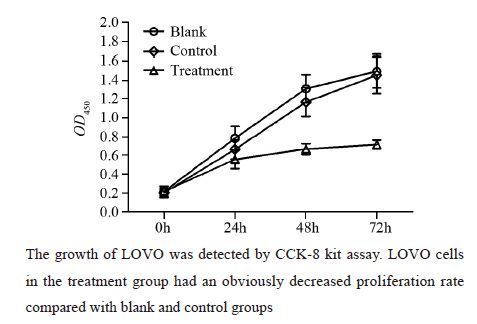
|
| 图 4 ATBF1过表达对LOVO增殖的抑制作用 Fig. 4 Overexpression of ATBF1 inhibited growth of LOVO cells |
如图 5所示,Q2、Q4象限,即FITC-C(+),表示早期凋亡的细胞。实验组LOVO细胞的早期凋亡率可达15.93%,而空白组和对照组的LOVO细胞早期凋亡率分别为5.23%和4.57%(P<0.001),表明细胞内ATBF1的高表达可导致LOVO的早期凋亡。

|
| 图 5 ATBF1过表达可诱导LOVO细胞凋亡 Fig. 5 Overexpression of ATBF1 induced apoptosis of LOVO cell |
小室上室的LOVO细胞穿过膜上Matrigel胶到膜的下室面,其迁移率的高低反映了肿瘤细胞侵袭能力的大小。收集转染48 h的细胞,采用Boyden小室检测三组细胞的侵袭情况。结果发现,与对照组、空白组比较,实验组的迁移率明显下降(P<0.001),见图 6。

|
| 图 6 ATBF1过表达可降低LOVO细胞侵袭性 Fig. 6 Overexpression of ATBF1 reduced invasion of LOVO cells |
2005年,ATBF1作为一个新发现的抑癌基因首次在前列腺癌的研究中被公开报道,在前列腺癌患者中,ATBF1基因突变率极高,而且突变的ATBF1主要发生在第8~10个外显子,突变后的ATBF1无法表达具有正常抑癌功能的ATBF1完整形式(即ATBF1-A异构体)[1, 10]。除了在前列腺癌,ATBF1在胃癌、乳腺癌、皮肤癌、头颈部鳞癌也均有研究[4, 6, 7, 8]。在胃癌的研究中发现,ATBF1-A的抑癌机制可能与其能与抑癌基因p53蛋白结合,形成ATBF1-p53复合体有关,这种复合体可产生2种调节基因转录的效果:(1)改变ATBF1-A的构象,促进ATBF1-A和AFP基因的增强子结合,抑制AFP基因转录;(2)促进p21基因的转录,加速癌细胞的凋亡进程[11]。在乳腺癌的研究中发现,ATBF1通过与AIB1竞争性结合到雌激素受体上,从而抑制雌激素介导的细胞增殖,同时还发现高浓度的雌激素可通过介导雌激素反应指蛋白(EFP),导致ATBF1蛋白的降解[12, 13, 14, 15]。
本研究中构建了ATBF1高表达的LOVO细胞模型,并在RT-PCR和Western blot中确认了该模型ATBF1的稳定表达性。Western blot结果显示,转染了pEGFP-ATBF1的LOVO细胞,即ATBF1高表达的实验组,Notch3蛋白的表达水平明显下调。研究表明,ATBF1能经转运进入癌细胞核内,与癌基因MUC5AC的AT模体元件结合,并抑制其转录调节功能[16],而MUC5AC能通过激活Notch3信号通路,引起胃肠肿瘤的发生[17]。实验结果与该结论相符,ATBF1高表达的LOVO细胞抑制了MUC5AC的活性,引起Notch3通路的失活,Notch3表达量下降,研究表明,Notch3通路的激活可以引起癌细胞的侵袭性增加与上皮间质转化[18]。这也意味着Notch3可能是ATBF1发挥抑癌作用时的一个下游因子。
在CCK-8增殖曲线可以看出,ATBF1高表达的LOVO细胞在转染72 h内,OD450值增长缓慢,表明细胞的增殖受到了抑制,显微镜下观察,pEGFP-ATBF1 LOVO细胞体积缩小,细胞质的量减少。与CCK-8的结果类似,Transwell实验也发现,实验组的迁移率明显低于其他两组,说明LOVO的侵袭性也同样受到了抑制。而以往研究发现,抑制ATBF1正常表达的前列腺癌细胞,增殖分化能力增加[19]。在我们之前对结直肠癌病例样本的研究就已经发现,已转移组的病理样本ATBF1表达量明显低于未转移组[9]。由此提示,由于ATBF1的缺失或突变,可增加结直肠癌细胞的转移和侵袭能力。
作为一种反式作用调节因子,ATBF1可促进多种癌细胞的凋亡。流式细胞术检测结果提示,转染pEGFP-ATBF1的LOVO细胞早期凋亡率明显高于空白组及对照组。研究表明,ATBF1的核定位可直接影响其蛋白表达[5],而ATBF1分子在核外转入核内的过程中,需要与RUNX3结合,并形成一个ATBF1-RUNX3-p53-smads复合体[20],而该复合体恰恰就是TGF-β信号通路中的重要一环,ATBF1的缺失,亦将导致复合体的失活,并最终无法介导癌细胞的凋亡。
本研究证实了结直肠癌细胞中ATBF1基因为调控侵袭和迁移的重要因子之一,作为一个抑癌基因,ATBF1对于肿瘤的发生发展起着关键作用。然而在其诱导肿瘤细胞凋亡,并进一步调控侵袭转移的过程,具体是通过何种信号通路和细胞因子协同作用的,仍然有待进一步探索。
| [1] | Sun X, Frierson HF, Chen C, et al. Frequent somatic mutations of the transcription factor ATBF1 in human prostate cancer[J]. Nat Genet, 2005, 37(4): 407-12. |
| [2] | Kataoka H, Miura Y, Joh T, et al. Alpha-fetoprotein producing gastric cancer lacks transcription factor ATBF1[J]. Oncogene, 2001, 20(7): 869-73. |
| [3] | Miura Y, Tam T, Ido A, et al. Cloning and characterization of an ATBF1 isoform that expresses in a neuronal differentiation-dependent manner[J]. J Biol Chem, 1995, 270(45): 26840-8. |
| [4] | Nishio E, Miura Y, Kawaguchi M, et al. Nuclear translocation of ATBF1 is a potential prognostic marker for skin cancer[J]. Acta Dermatovenerol Croat, 2012, 20(4): 239-45. |
| [5] | Sun X, Li J, Dong FN, et al. Characterization of nuclear localization and SUMOylation of the ATBF1 transcription factor in epithelial cells[J]. PLoS One, 2014, 9(3): e92746. |
| [6] | Sun X, Zhou Y, Otto KB, et al. Infrequent mutation of ATBF1 in human breast cancer[J]. J Cancer Res Clin Oncol, 2007, 133(2): 103-5. |
| [7] | Lew DH, Jung WT, Kim HJ, et al. Clinicopathological characteristics and prognosis of alpha-fetoprotein producing gastric cancer[J]. Korean J Gastroenterol, 2013, 62(6): 327-35. |
| [8] | Sun X, Li J, Sica G, et al. Interruption of nuclear localization of ATBF1 during the histopathologic progression of head and neck squamous cell carcinoma[J]. Head Neck, 2013, 35(7): 1007-14. |
| [9] | Zhong L, Chen F, Li Q, et al. ATBF1 expression and correlation with metastasis in colorectal cancer[J]. Kun Ming Yi Ke Da Xue Xue Bao, 2014, 35(6): 37-41. [钟林, 陈飞, 李强, 等. ATBF1在结直肠癌中的表达及其与转移相关性研究[J]. 昆明医科大学学报, 2014, 35(6): 37-41.] |
| [10] | Xu J, Sauvageot J, Ewing CM, et al. Germline ATBF1 mutations and prostate cancer risk[J]. Prostate, 2006, 66(10): 1082-5. |
| [11] | Miura Y, Kataoka H, Joh T, et al. Susceptibility to killer T cells of gastric cancer cells enhanced by Mitomycin-C involves induction of ATBF1 and activation of p21 (Waf1/Cip1) promoter[J]. Microbiol Immunol, 2004, 48(2): 137-45. |
| [12] | Dong XY, Fu X, Fan S, et al. Oestrogen causes ATBF1 protein degradation through the oestrogen-responsive E3 ubiquitin ligase EFP[J]. Biochem J, 2012, 444(3): 581-90. |
| [13] | Dong XY, Guo P, Sun X, et al. Estrogen up-regulates ATBF1 transcription but causes its protein degradation in estrogen receptor-alpha-positive breast cancer cells[J]. J Biol Chem, 2011, 286(16): 13879-90. |
| [14] | Dong XY, Sun X, Guo P, et al. ATBF1 inhibits estrogen receptor (ER) function by selectively competing with AIB1 for binding to the ER in ER-positive breast cancer cells[J]. J Biol Chem, 2010, 285(43): 32801-9. |
| [15] | Li M, Zhao D, Ma G, et al. Upregulation of ATBF1 by progesterone-PR signaling and its functional implication in mammary epithelial cells[J]. Biochem Biophys Res Commun, 2013, 430(1): 358-63. |
| [16] | Mori Y, Kataoka H, Miura Y, et al. Subcellular localization of ATBF1 regulates MUC5AC transcription in gastric cancer[J]. Int J Cancer, 2007, 121(2): 241-7. |
| [17] | Eom DW, Hong SM, Kim J, et al. Notch3 signaling is associated with MUC5AC expression and favorable prognosis in patients with small intestinal adenocarcinomas[J]. Pathol Res Pract, 2014, 210(8): 501-7. |
| [18] | Li C, Song G, Zhang S, et al. Wnt3a increases the metastatic potential of non-small cell lung cancer cells in vitro in part via its upregulation of Notch3[J]. Oncol Rep, 2015, 33(3): 1207-14. |
| [19] | Sun X, Fu X, Li J, et al. Deletion of atbf1/zfhx3 in mouse prostate causes neoplastic lesions, likely by attenuation of membrane and secretory proteins and multiple signaling pathways[J]. Neoplasia, 2014, 16(5): 377-89. |
| [20] | Mabuchi M, Kataoka H, Miura Y, et al. Tumor suppressor, AT motif binding factor 1 (ATBF1), translocates to the nucleus with runt domain transcription factor 3 (RUNX3) in response to TGF-beta signal transduction[J]. Biochem Biophys Res Commun, 2010, 398(2): 321-5. |
 2016, Vol. 43 No.4
2016, Vol. 43 No.4