文章信息
- 刘娟, 燕莞瑶, 楼一层.
- LIU Juan, YAN Wanyao, LOU Yiceng.
- NOTCH1对T-ALL抑癌基因ID4的负调控作用
- NOTCH1 Down-regulates Tumor Suppressor Gene ID4 in T-cell Acute Lymphoblastic Leukemia
- 肿瘤防治研究, 2016, 43(02): 123-128
- Cancer Research on Prevention and Treatment, 2016, 43(02): 123-128
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2016.02.006
-
文章历史
- 收稿日期: 2015-09-08
- 修回日期: 2015-12-17
2. 430030 武汉,华中科技大学同济医学院药学院
2. School of Pharmacy,Tongji Medical College,Huazhong University of Science and Technology,Wuhan 430030,China
急性T淋巴细胞白血病(T-cell acute lymphoblastic leukemia,T-ALL)是一类严重威胁人类健康的血液系统恶性疾患,在儿童及成人发病率较高。目前主要采取联合化疗来治疗T-ALL[1, 2],但患者死亡率仍很高而且容易复发。因此迫切需要新的治疗策略提高T-ALL的生存率。
NOTCH信号通路与肿瘤的关系最早发现于急性T淋巴细胞白血病[3]。T-ALL发生时NOTCH会被异常地和组成性地激活[4, 5],NOTCH信号通路的激活释放出NOTCH的细胞内肽段/细胞内活化形式(intracellular notch,ICN)[6, 7],ICN在造血干细胞中表达的增加引起T-ALL。有研究报道,大约有55% T-ALL患者带有NOTCH1获得性功能突变,说明NOTCH1的异常激活是T-ALL的重要发病机制。γ分泌酶抑制剂Compound E(GSI)能够有效抑制NOTCH1的激活,但GSI作用不专一,除了阻断NOTCH1信号通路的激活,同时还阻断了其他NOTCH信号通路,出现明显不良反应。因此,寻找靶向NOTCH1下游靶基因的药物是今后T-ALL治疗的重要方向。HES1作为已发现的直接受NOTCH1信号通路正调控的基因,对NOTCH1信号通路过度激活所致T-ALL的发生与维持具有至关重要的作用,HES1的表达水平反映NOTCH1的细胞内活化形式ICN1的活性[8, 9],因而HES1作为NOTCH1信号通路效应分子。
微小RNA(microRNA,miRNA)是长度20~24 nt的非编码小RNA,通过与mRNA 特异性结合实行转录后负性调节基因表达。新近研究发现miRNA确定在T淋巴细胞分化和发展的各阶段发挥着重要的生理功能,并且参与了各类癌症包括T-ALL的形成和恶性发展[10, 11, 12]。
DNA结合抑制因子4(inhibitor of DNA binding 4,ID4)与肿瘤的发生和干细胞的分化关系密切,该基因的表达缺失与多种急性白血病的发生密切相关。在多种白血病细胞中沉默ID4基因会促进白血病的发生,因而ID4基因是一个明确的抗白血病基因[13, 14],但是调控机制仍然不十分明确。本研究旨在探索ID4在T-ALL中的表达情况以及NOTCH1与ID4的相关性,并深入探究NOTCH1与ID4之间的调控机制,为遏制T-ALL发生发展提供新的可行方案。
1 材料和方法 1.1 试剂胎牛血清购自美国Gibco公司;RPMI 1640培养液购自美国Hyclone公司;细胞转染试剂盒V购自瑞士Lonza公司;γ分泌酶抑制剂Compound E(GSI)购自德国Merck公司;二甲基亚砜(DMSO)购自美国Sigma公司;氯仿、无水乙醇和异丙醇购自天津天力化学试剂有限公司;Trizol、miRNA分离试剂盒和miRNA反转录试剂盒均购自美国Life Technologies公司;反转录试剂盒购自美国Thermo Fisher Scientific公司;SYBR Green购自美国BIO-RAD公司;RIPA裂解液购自上海Beyotime公司;ID4一抗(兔源多克隆抗体)购自英国Abcam公司;HRP标记的二抗购自美国Cell Signaling公司;PGL3质粒及Dual-luciferase检测试剂盒购自美国Promega公司;miR-342抑制体(miR-342 inhibitor)、miR-342抑制体阴性对照(无关序列,miR-342 inhibitor NC)、miR-342模拟物(miR-342 mimics)及miR-342模拟物阴性对照(无关序列,miR-342 mimics NC)均由上海吉玛生物公司设计合成。
1.2 细胞株与细胞培养人源急性T淋巴细胞白血病细胞株Jurkat细胞、PCDH-ICN质粒包装慢病毒感染后经嘌呤霉素筛选得到过表达ICN的Jurkat细胞及与其配对的对照组PCDH质粒包装慢病毒感染后经嘌呤霉素筛选得到的Jurkat细胞均来源于Warren S Pear实验室。其培养液是含有10%小牛血清、2 mmol/L谷氨酰氨、1 mmol/L丙酮酸钠、非必需氨基酸和β-巯基乙醇的RPMI 1640。5%CO2、37℃培养。
1.3 生物信息学miRNA靶基因预测使用目前公认的miRNA靶基因预测数据库TargetScan、picTar、RNA22、PITA和miRanda共同预测可以靶向ID4的miRNA。对将预测出来的miRNA序列与靶基因ID4 3’UTR进行碱基互补比对并计算结合稳定性。
1.4 双荧光素酶报告系统验证为证明miR-342特异作用于ID4 3’UTR区域,构建含有ID4 mRNA的3’UTR(PGL3/ID4)及其3’UTR突变体(PGL3/mut)的双荧光报告基因系统。方法是针对ID4 3’UTR预测结合miR-342区域,设计PCR引物,将PCR产物连在骨架质粒PGL3中海肾(Renilla)的下游,导入感受态细胞后摇菌,进行克隆筛选后送苏州金唯智生物科技有限公司基因测序,结果经比对正确后再摇菌小提得到目的质粒PGL3 /ID4。在Jurkat细胞中共转染miR-342 inhibitor、miR-342 mimics和PGL3/ID4,只转PGL3 /ID4作为对照,24 h后使用Dual-luciferase检测试剂盒读取萤火虫(firefly)信号和海肾(Renilla)荧光信号,以Rellina/firefly相对荧光值作比较。对确认有效的ID4 3’UTR区域,利用重叠PCR原理设计引物制造结合区域点突变,余过程如上。经苏州金唯智生物科技有限公司测序明确的miR结合区域点突变后,提取得到PGL3/mut,共转染验证是否miR-342还能结合并调控Renilla表达。
1.5 核转染(nucleofection)实验根据Lonza Cell Line Nucleofector Kit V 试剂盒说明书,用核转染仪4D-Nucleofector(Lonza)分别将miR-342 inhibitor、miR-342 inhibitor NC、miR-342 mimics及miR-342 mimics NC转入Jurkat细胞,方法如下: 细胞计数仪计数细胞,锥虫蓝拒染实验检测细胞存活率后将1×106 细胞用1.5 ml RPMI 1640完全培养液接种于12孔培养板,37℃、5%CO2培养箱中预热。细胞移至1.5 ml EP管中,室温下800 r/min离心5 min,弃上清液,细胞用100 μl预热到室温的核转染液(Nucleofector Solution 82 μl+Supplement 18 μl)混悬,并加入30 nmol/L miR-342 inhibitor/miR-342 inhibitor NC/miR-342 mimics/miR-342 mimi cs NC,转移到Lonza核转染杯的底部,避免产生气泡,盖上杯盖。将核转染仪的程序设置为CL-120,并将核转染杯放入杯孔中,按“X”键开始程序。结束后立即拿出转染杯,并用Lonza标准吸管吸出移至含有1.5 ml完全培养液的12孔板中。48 h后收集转染后的细胞,进行后续实验。
1.6 总RNA的提取及qRT-PCR检测利用Trizol提取细胞总RNA,取1 μg总RNA做反转录反应。PCR扩增程序如下: 50℃ 2 min,95℃ 10 min,95℃ 15 s,60℃ 1 min,共进行40个循环。PCR引物分别为: 18 s上游引物:5′-GCGCCGCTAGAGGTGAAAT-3′,下游引物:5′-GGCGGGTCATGGGAATAAC-3′;HES1上游引物:5′-TCAACACGACACCGGATAAA-3′,下游引物:5′-TCAGCTGGCTCAAGACTTTCA-3′;ID4上游引物:5′-TGAACAAGCAGGGCGACAG-3′,下游引物:5′-CCCTCTCTAGTGCTCCTGGCT-3′。
1.7 miRNA的提取及qRT-PCR检测利用miRNA分离试剂盒提取细胞miRNA,取1 μg miRNA利用miRNA反转录试剂盒做反转录反应。PCR扩增程序如下: 50℃ 2 min,95℃ 10 min,95℃ 15 s,60℃ 1 min,共进行40个循环。miR-342的特异性反转录引物及PCR引物均由上海吉玛生物公司设计合成。
1.8 Western blot检测ID4蛋白表达水平收集DMSO处理组、GSI处理组及不同转染组的Jurkat细胞,加入蛋白裂解液,BCA法测定蛋白浓度。取50 μg样品蛋白质加入等体积上样缓冲液,经SDS-PAGE后,用水浴式电转仪转至硝酸纤维素膜上。室温封闭1 h,加入1:1 000稀释的ID4一抗,4℃孵育过夜,β-actin作为内参照,TBST振摇漂洗5 min×3次,然后加入HRP标记的二抗,室温孵育1 h,取出,TBST 振摇漂洗5 min×3次,化学发光法检测后分析结果,结果用Image J软件分析条带灰度值。
1.9 统计学方法采用SPSS 17.0统计软件处理数据,以均数±标准差(x±s)表示,均数间的比较采用t检验或方差分析,P<0.05为差异具有统计学意义。
2 结果 2.1 ID4在T-ALL患者中低表达根据Oncomine数据库,ID4在T-ALL患者(174例)中的表达量为正常捐献者(74例)的(0.425±0.021)倍(P<0.01),结果表明T-ALL患者中ID4的表达水平明显低于正常组,见图 1。

|
| **: P<0.01,compared with normal donors; T-ALL: T-cell acute lymphoblastic leukemia; ID4: inhibitor of DNA binding 4 图 1 Oncomine数据库中ID4在T-ALL患者与正常组中表达水平的分析结果比较 Fig. 1 Comparison of ID4 expression levels in 174 T-ALL patients and 74 normal donors from Oncomine database |
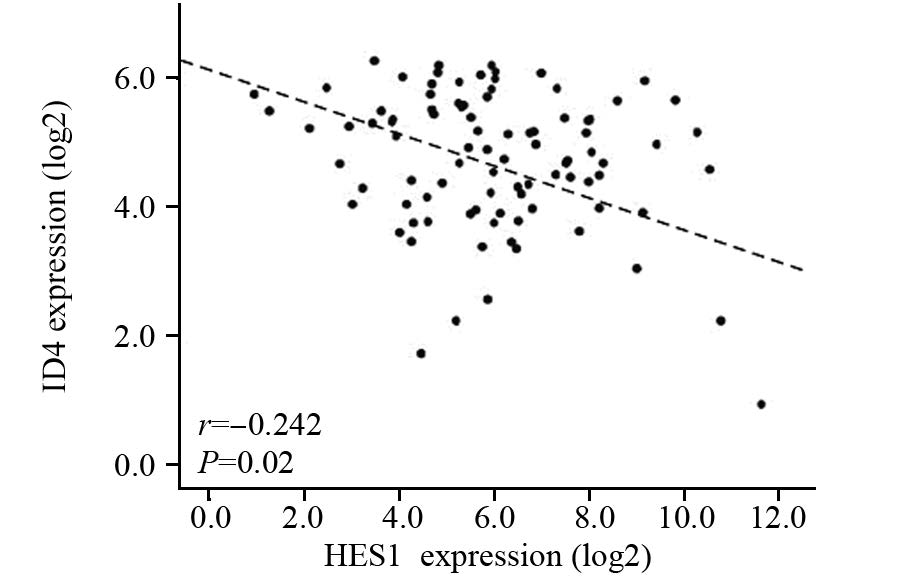
推测T-ALL中ID4的低表达可能与NOTCH1的过度激活有关。通过分析源于Pieters R数据库的92例T-ALL患者样本中HES1与ID4表达水平的相关性(http://r2.amc.nl,last accessed August 20,2015),可知HES1与ID4呈负相关(r=-0.242,P=0.02),见图 2,由此间接表明ID4可能受NOTCH1负调控。
|
| 图 2 源于Pieters R数据库的92例T-ALL患者中HES1与ID4表达水平的相关性 Fig. 2 Correlation between HES1 and ID4 expression levels of 92 T-ALL patients from Pieters R database |
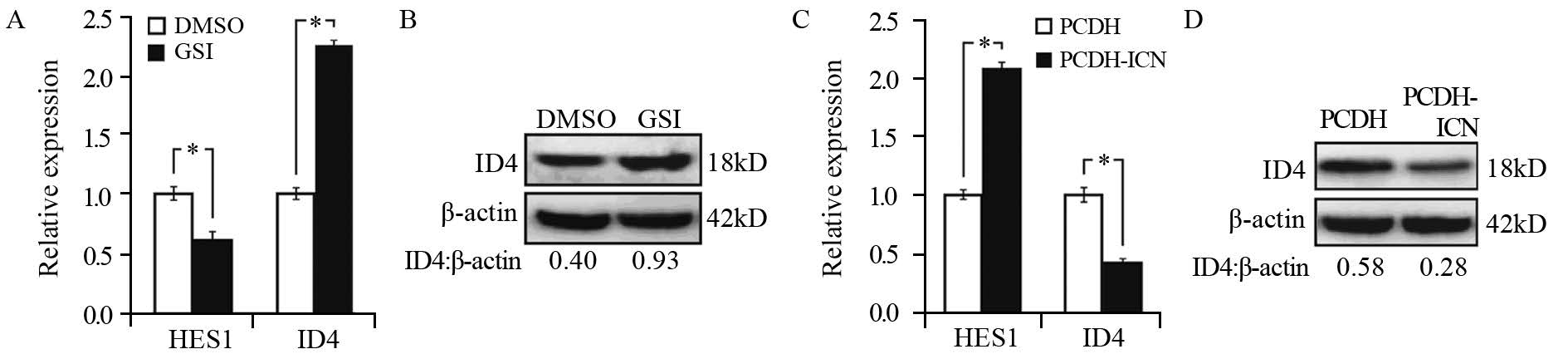
将Jurkat细胞分别用1 μmol/L可阻断NOTCH1信号通路激活的γ分泌酶抑制剂Compound E(GSI)和空白对照DMSO处理48 h,qRT-PCR结果显示,与DMSO空白对照组相比,GSI组的ID4 mRNA表达水平上调至223%(t=30.1,P=0.001),见图 3A;Western blot法检测和灰度结果分析显示GSI组的ID4表达上调至233%(t=28.8,P=0.001),见图 3B。
|
| A: qRT-PCR results of HES1 and ID4 mRNA expression in Jurkat cells with GSI-mediated NOTCH1 inhibition; B: Western blot results of ID4 protein expression in Jurkat cells with GSI-mediated NOTCH1 inhibition; C: qRT-PCR results of HES1 and ID4 mRNA expression in Jurkat cells with PCDH-ICN-mediated NOTCH1 activation; D: Western blot results of ID4 protein expression in Jurkat cells with PCDH-ICN-mediated NOTCH1 activation; *: P<0.05 /span> 图 3 阻断或激活NOTCH1信号通路后,Jurkat细胞中ID4的mRNA和蛋白水平表达 Fig. 3 ID4 mRNA and protein expression in Jurkat cells with GSI-mediated NOTCH1 inhibition or PCDH-ICN-mediated NOTCH1 activation |
qRT-PCR和Western blot法检测已转染PCDH-ICN能激活NOTCH1信号通路及其对照PCDH组的Jurkat细胞中ID4的表达,结果显示,与PCDH对照组相比,PCDH-ICN组的ID4 mRNA表达水平下调至43%(t=19.3,P=0.000),见图 3C;ID4蛋白表达水平下调至48%(t=9.2,P=0.000),见图 3D。
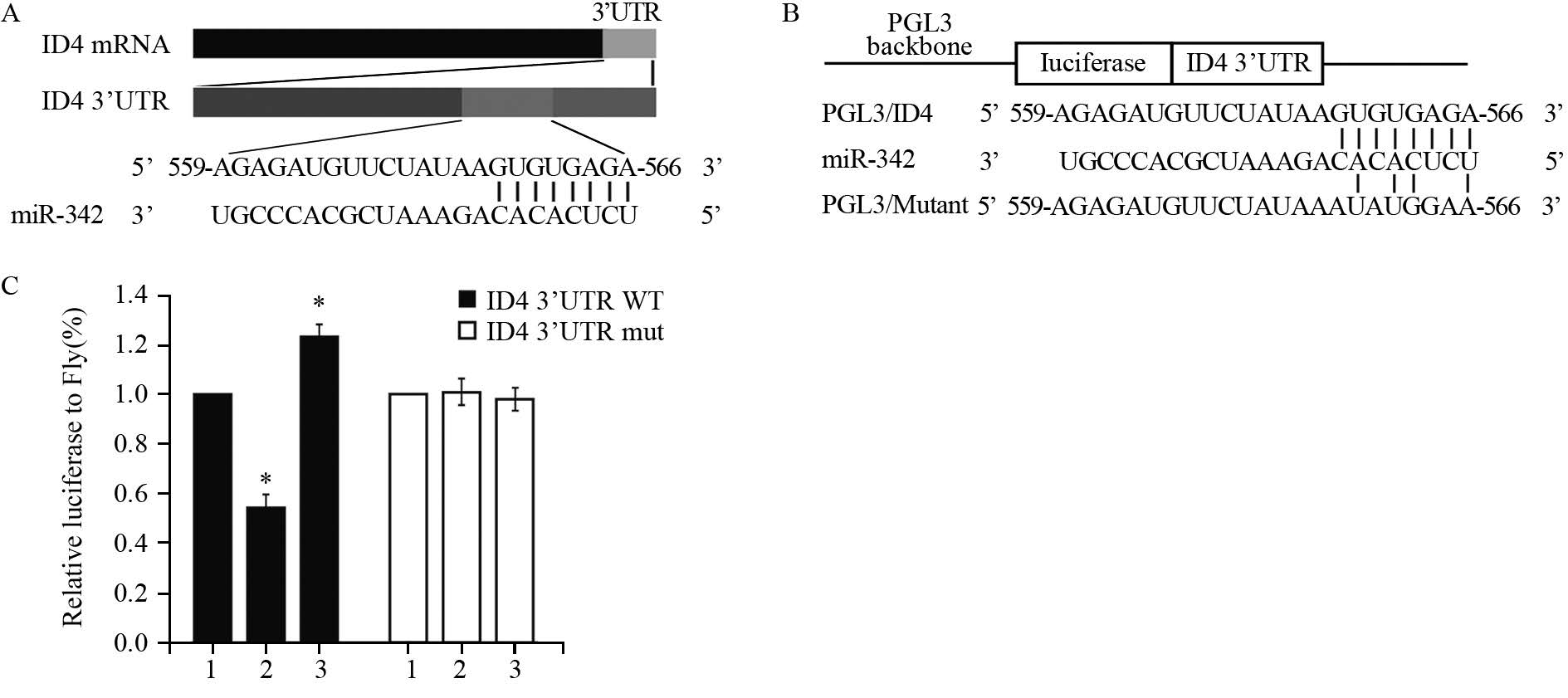
2.4 miR-342直接靶向ID4通过目前公认的miRNA靶基因预测数据库TargetScan、picTar、RNA22、PITA和miRanda共同预测到只有miR-342可以结合于ID4 mRNA 3’UTR区的第559~566号碱基,见图 4A。
|
| A: the binding site of ID4 mRNA 3’UTR predicted by TargetScan,picTar,RNA22,PITA and miRanda algorithms analysis; B: Schematic representation of the reporter for miR-342 activity against the ID4-3’UTR; C: miR-342 mimics and inhibitor caused specific contrary effect towards ID4-3’UTR after using the reporter system in Jurkat cells (P<0.05),but no significant effect in the ID4-3’UTR-mutant(P>0.05); *: P<0.05,compared with control group; 1: Control; 2: miR-342 mimics; 3: miR-342 inhibitor 图 4 ID4 mRNA的3’UTR区与miR-342相结合 Fig. 4 miR-342 directly targeted ID4 by interacting with its 3’UTR |
构建含有ID4 mRNA 3’UTR的双荧光素酶报告载体,称为PGL3/ID4,见图 4B,结果表明当PGL3/ID4组与miR-342 mimics共转染后,较对照组海肾相对荧光(Renilla/firefly)减少到58%(t=11.4,P=0.000);与miR-342 inhibitor共转染后,海肾相对荧光升高到125%(t=15.7,P=0.001);而PGL3/mut分别与miR-342 mimics或miR-342 inhibitor共转染后海肾相对荧光基本无改变(P>0.05),见图 4C,提示miR-342可以与ID4的3’UTR区相结合。
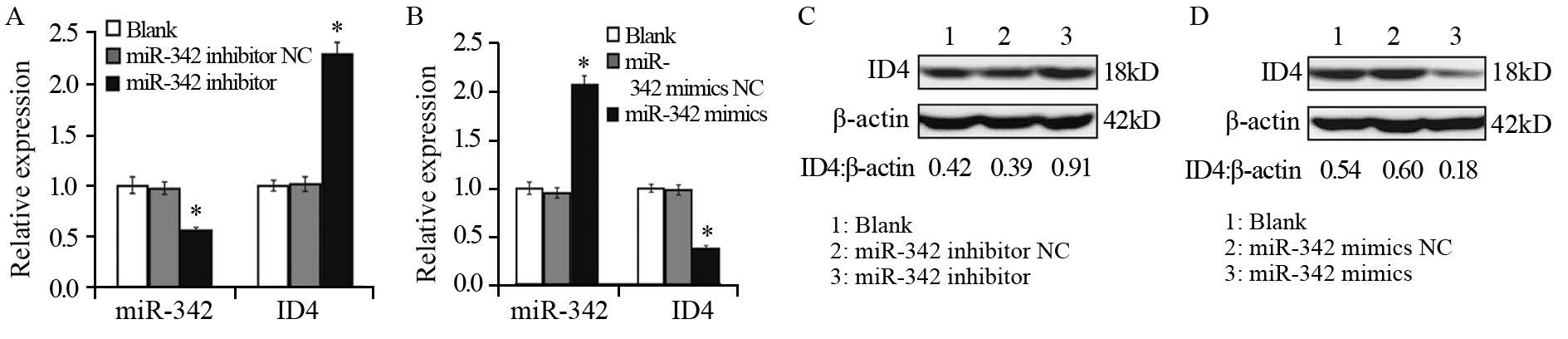
2.5 miR-342调控ID4 mRNA和蛋白的表达将miR-342 inhibitor NC、miR-342 inhibitor、miR-342 mimics NC及miR-342 mimics转染Jurkat细胞48 h,检测细胞中ID4的mRNA及蛋白表达水平。qRT-PCR结果显示,与miR-342 inhibitor NC对照组相比,miR-342 inhibitor组ID4 mRNA表达水平上调至225%(t=21.5,P=0.000),见图 5A;与miR-342 mimics NC对照组相比,miR-342 mimics组的ID4 mRNA表达水平下调至37%(t=19.4,P=0.001),见图 5B。Western blot法检测和灰度结果分析显示miR-342 inhibitor组的ID4表达相比miR-342 inhibitor NC对照组上调至233%(t=10.7,P=0.001),见图 5C;miR-342 mimics组的ID4表达相比miR-342 mimics NC对照组下调至30%(t=18.4,P=0.000),见图 5D。
|
| A: qRT-PCR results of miR-342 and ID4 expression in Jurkat cells transfected with miR-342 inhibitor NC or miR-342 inhibitor,*: P<0.05,compared with miR-342 inhibitor NC or blank group; B: qRT-PCR results of miR-342 and ID4 expression in Jurkat cells transfected with miR-342 mimics NC or miR-342 mimics,*: P<0.05,compared with miR-342 mimics NC or blank group; C: Western blot results of ID4 protein expression in Jurkat cells transfected with miR-342 inhibitor NC or miR-342 inhibitor; D: Western blot results of ID4 protein expression in Jurkat cells transfected with miR-342 mimics NC or miR-342 mimics 图 5 下调miR-342或上调miR-342、Jurkat细胞中ID4的mRNA和蛋白水平表达 Fig. 5 ID4 mRNA and protein expression in Jurkat cells after down- or up-regulation of miR-342 |
对Jurkat细胞分别用1 μmol/L GSI和DMSO处理24 h,再将miR-342 mimics分别转染上述GSI和DMSO处理组细胞48 h,检测DMSO空白对照组、GSI处理组和GSI+miR-342 mimics转染组的细胞中miR-342的表达和ID4的mRNA及蛋白表达水平。qRT-PCR结果显示,与DMSO空白对照组相比,GSI处理组的miR-342的表达下调至59%(t=19.8,P=0.000),ID4 mRNA表达水平上调至189%(t=27.4,P=0.001);GSI+miR-342 mimics转染组的miR-342的表达仅下调至91%(t=17.2,P=0.001),ID4 mRNA表达水平仅上调至130%(t=25.4,P=0.000),见图 6A;Western blot法检测和灰度结果分析显示GSI处理组相比DMSO空白对照组ID4表达上调至258%(t=8.9,P=0.000),而GSI+miR-342 mimics转染组相比DMSO空白对照组ID4表达仅上调至165%(t=12.7,P=0.001),见图 6B。
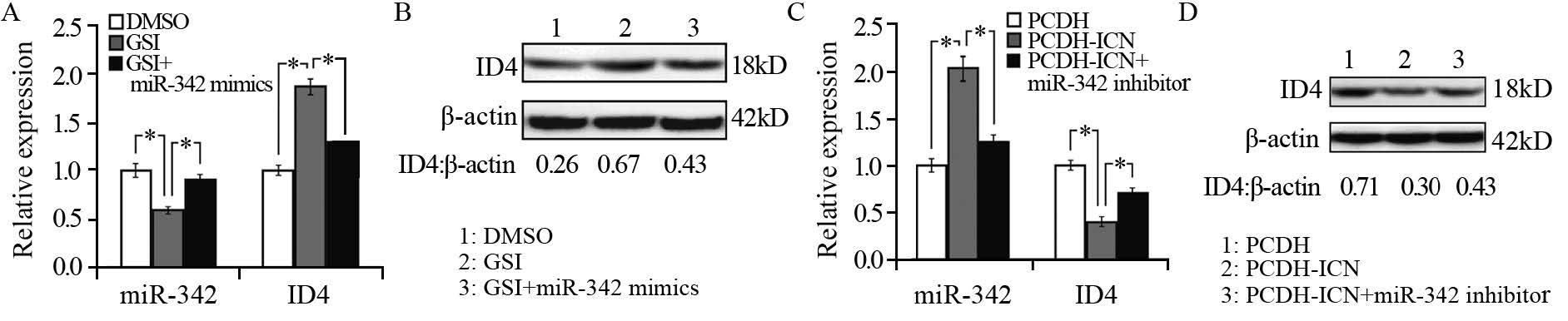
|
| A: qRT-PCR results of miR-342 and ID4 expression in Jurkat cells with GSI-mediated NOTCH1 inhibition or miR-342 mimics-transfected Jurkat cells treated with GSI for 24h; B: Western blot results of ID4 protein expression in Jurkat cells with GSI-mediated NOTCH1 inhibition or miR-342 mimics-transfected Jurkat cells treated with GSI for 24h; C: qRT-PCR results of miR-342 and ID4 expression in Jurkat cells with PCDH-ICN-mediated NOTCH1 activation or miR-342 inhibitor-transfected Jurkat cells with PCDH-ICN-mediated NOTCH1 activation; D: Western blot results of ID4 protein expression in Jurkat cells with PCDH-ICN-mediated NOTCH1 activation or miR-342 inhibitor-transfected Jurkat cells with PCDH-ICN-mediated NOTCH1 activation; *: P<0.05 图 6 miR-342介导NOTCH1对ID4 mRNA和蛋白水平表达的负调控作用 Fig. 6 miR-342 mediated negative regulation effect of NOTCH1 on ID4 mRNA and protein expression |
将miR-342 inhibitor分别转染上述已转染PCDH和PCDH-ICN的Jurkat细胞48 h,检测PCDH对照组、PCDH-ICN组和PCDH-ICN+miR-342 inhibitor转染组的细胞中miR-342的表达和ID4的mRNA及蛋白表达水平。qRT-PCR结果显示,与PCDH对照组相比,PCDH-ICN组的miR-342的表达上调至204%(t=29.4,P=0.001),ID4 mRNA表达水平下调至40%(t=31.7,P=0.000);PCDH-ICN+miR-342 inhibitor转染组的miR-342的表达仅上调至126%(t=16.9,P=0.000),ID4 mRNA表达水平仅下调至71%(t=24.3,P=0.000),见图 6C;Western blot法检测和灰度结果分析显示PCDH-ICN组相比PCDH对照组ID4表达下调至42%(t=10.6,P=0.001),而PCDH-ICN+miR-342 inhibitor转染组相比PCDH对照组ID4表达仅下调至61%(t=13.7,P=0.000),见图 6D。
3 讨论大部分T-ALL患者都会发生NOTCH信号通路的激活突变,这表明异常激活的NOTCH信号通路在该病的发生中发挥了重要作用。NOTCH信号通路通过调节靶基因的表达来调节致癌或抑癌基因信号,而且有研究表明microRNAs (miRNAs)也参与了NOTCH调节靶基因表达的过程[15, 16]。因此针对介导NOTCH调控靶基因的miRNA的深入探索,对于揭示T-ALL发生的分子机制和研究抗T-ALL药物靶点有重要意义。
本研究发现了ID4在T-ALL患者中低表达,并且NOTCH1负调控ID4。通过生物信息学方法发现miR-342直接靶向ID4,接着通过构建双荧光素酶报告系统验证了miR-342特异结合ID4 3’-UTR区域调控基因表达,上调miR-342后mRNA和蛋白水平检测ID4显著下调,下调miR-342后mRNA和蛋白水平检测ID4显著上调。结合李三中[17]报道的基因芯片结果,即以构建特异性NOTCH阻断小鼠为基础,通过高通量筛选出miR-342是NOTCH信号下游的microRNA之一,我们推测在T-ALL中,NOTCH1信号通路可能参与了miR-342的调控。
本实验证实了上面的推测,在T-ALL中阻断NOTCH1信号通路后,与对照组相比,miR-342表达下调,ID4 mRNA和蛋白水平均上调;激活NOTCH1信号通路后,与对照组相比,miR-342表达上调,ID4 mRNA和蛋白水平均下调,这说明NOTCH1信号通路在T-ALL中可激活 miR-342的表达。综上所述,本研究证明了NOTCH1通过激活miR-342对ID4的负调控作用从而下调抑癌基因ID4的表达,促进T-ALL的发生。并可以进一步推测,miR-342的表达高低对T-ALL的发生具有重要影响,miR-342可能作为新的生物学标志用于肿瘤诊断,其促进T-ALL发生的特性,为抗T-ALL新药的研发提供理论基础。
miR-342在多种肿瘤中被证实为肿瘤相关miRNA,可以影响不同的肿瘤细胞的生长、转移或耐药等。Xie等[18]报道了miR-342通过调控RAP2B参与非小细胞肺癌细胞系的生长和侵袭;在宫颈癌中,也有报道miR-342通过靶向FOXM1抑制宫颈癌细胞的生长、转移和侵袭[19];He等[20]发现了miR-342能通过调控雌激素受体参与到乳腺癌的化疗耐药过程。由于miRNA和其靶基因作用关系网络复杂,一个miRNA可以调控多个靶基因,一个靶基因可以被多个miRNA调控,形成复杂的反馈调节关系,因此推测miR-342不仅可以介导NOTCH1调控ID4从而下调抑癌基因ID4的表达的过程,还有可能通过其他的作用靶点来影响T-ALL细胞的生长、耐药等恶性生物学行为。如何更全面地阐明与T-ALL相关的miRNA在T-ALL发生发展中的作用和介导NOTCH1调 控 靶基因复杂的调控网络,仍需进一步探索。
| [1] | Tosello V, Ferrando AA. The NOTCH signaling pathway: role in the pathogenesis of T-cell acute lymphoblastic leukemia and implication for therapy[J]. Ther Adv Hematol, 2013, 4(3): 199-210. |
| [2] | Roti G, Stegmaier K. New Approaches to Target T-ALL[J]. Front Oncol, 2014, 4: 170. |
| [3] | Aster JC, Blacklow SC, Pear WS. Notch signalling in T-cell lymphoblastic leukaemia/lymphoma and other haematological malignancies[J]. J Pathol, 2011, 223(2): 262-73. |
| [4] | Paganin M, Ferrando AA. Molecular pathogenesis and targeted therapies for NOTCH1-induced T-cell acute lymphoblastic leukemia[J]. Blood Rev, 2011, 25(2): 83-90. |
| [5] | Tatarek J, Cullion K, Ashworth T, et al. Notch1 inhibition targets the leukemia-initiating cells in a Tal1/Lmo2 mouse model of T-ALL[J]. Blood, 2011, 118(6): 1579-90. |
| [6] | Arnett KL, Hass M, McArthur DG, et al. Structural and mechanistic insights into cooperative assembly of dimeric Notch transcription complexes[J]. Nat Struct Mol Biol, 2010, 17(11): 1312-7. |
| [7] | Zage PE, Nolo R, Fang W, et al. Notch pathway activation induces neuroblastoma tumor cell growth arrest[J]. Pediatr Blood Cancer, 2012, 58(5): 682-9. |
| [8] | Wendorff AA, Koch U, Wunderlich FT, et al. Hes1 is a critical but context-dependent mediator of canonical Notch signaling in lymphocyte development and transformation[J]. Immunity, 2010, 33(5): 671-84. |
| [9] | Kannan S, Fang W, Song G, et al. Notch/HES1-mediated PARP1 activation: a cell type-specific mechanism for tumor suppression[J]. Blood, 2011, 117(10): 2891-900. |
| [10] | Jernås M, Nookaew I, Wadenvik H, et al. MicroRNA regulate immunological pathways in T-cells in immune thrombocytopenia (ITP)[J]. Blood, 2013, 121(11): 2095-8. |
| [11] | Iorio MV, Croce CM. MicroRNA dysregulation in cancer: diagnostics, monitoring and therapeutics. A comprehensive review[J]. EMBO Mol Med, 2012, 4(3): 143-59. |
| [12] | Mavrakis KJ, Van Der Meulen J, Wolfe AL, et al. A cooperative microRNA-tumor suppressor gene network in acute T-cell lymphoblastic leukemia (T-ALL)[J]. Nat Genet, 2011, 43(7): 673-8. |
| [13] | Chen SS, Claus R, Lucas DM, et al. Silencing of the inhibitor of DNA binding protein 4 (ID4) contributes to the pathogenesis of mouse and human CLL[J]. Blood, 2011, 117(3): 862-71. |
| [14] | Hu HB, Hu Q. ID4 methylation patterns in childhood T line and B line lymphocytic leukemia[J]. Zhongguo Dang Dai Er Ke Za Zhi, 2010, 12: 940-2. [胡洪玻, 胡群. T系和B系急性淋巴细胞白血病ID4甲基化状态分析[J]. 中国当代儿科杂志, 2010, 12: 940-2.] |
| [15] | Prokopi M, Kousparou CA, Epenetos AA. The Secret Role of microRNAs in Cancer Stem Cell Development and Potential Therapy: A Notch-Pathway Approach[J]. Front Oncol, 2014, 4:389. |
| [16] | Da Ros VG, Gutierrez-Perez I, Ferres-Marco D, et al. Dampening the signals transduced through hedgehog via microRNA miR-7 facilitates notch-induced tumourigenesis[J]. PLoS Biol, 2013, 11(5): e1001554. |
| [17] | Li SZ. The role of Notch signaling pathway in the mechanism of glioma pathogenesis[D]. Di Si Jun Yi Da Xue, 2013. [李三中. Notch信号途径在胶质瘤发病机制中的作用研究[D]. 第四军医大学, 2013.] |
| [18] | Xie X, Liu H, Wang M, et al. miR-342-3p targets RAP2B to suppress proliferation and invasion of non-small cell lung cancer cells[J]. Tumour Biol, 2015, 36(7): 5031-8. |
| [19] | Li XR, Chu HJ, Lv T, et al. miR-342-3p suppresses proliferation, migration and invasion by targeting FOXM1 in human cervical cancer[J]. FEBS Lett, 2014, 588(17): 3298-307. |
| [20] | He YJ, Wu JZ, Ji MH, et al. miR-342 is associated with estrogen receptor-alpha expression and response to tamoxifen in breast cancer[J]. Exp Ther Med, 2013, 5(3): 813-8. |
 2016, Vol. 43
2016, Vol. 43


