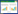文章信息
- 李伟,朱明月,董栩,夏华,陈栘,鲁琰,郭峻莉,谢协驹,符史干,李孟森. 2015.
- LI Wei, ZHU Mingyue, DONG Xu, XIA Hua, CHEN Yi, LU Yan, GUO Junli1, XIE Xieju1, FU Shigan, LI Mengsen. 2015.
- 曲古抑菌素A增加人肝癌Bel7402细胞对TRAIL敏感度的实验
- Trichostatin A Increases Sensitivity of Hepatocellular Carcinoma Cells Bel7402 to TRAIL
- 肿瘤防治研究, 2015, 42(06): 541-547
- Cancer Research on Prevention and Treatment, 2015, 42 (06): 541-547
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2014.09.002
-
文章历史
- 收稿日期:2014-06-13
- 修回日期:2014-12-26
2. 571199 海口,海南医学院分子生物学重点实验室;
3. 571199 海口,海南医学院病理生理学教研室;
4. 571199 海口,海南医学院生理学教研室;
5. 570102 海口,海南医学院肿瘤研究所
2. Hainan Key Laboratory of Molecular Biology, Hainan Medical College, Haikou 571199, China;
3. Department of Pathophysiology, Hainan Medical College, Haikou 571199, China;
4. Department of Physiology, Hainan Medical College, Haikou 571199, China;
5. Institution of Tumor, Hainan Medical College, Haikou 570102, China
肝癌严重威胁人类健康,目前治疗主要是放化疗、手术切除和移植。临床上对肝癌的治疗多采取化学药物治疗,但在治疗的过程中,肝癌细胞耐药是一直难以突破的问题,因而多药联合使用是一种有效的治疗方法和策略。肿瘤坏死因子(tumor necrosis factor,TNF)相关凋亡诱导配体(TNF related apoptosis inducing ligand,TRAIL)是Wiley等[1]首次从人心肌cDNA文库中克隆,其能与死亡受体结合,启动凋亡信号转导途径,诱导肿瘤细胞凋亡,而对正常组织和细胞没有明显的毒副作用[2],因而是一个非常有开发前景的治疗肿瘤的细胞因子[3]。但是,很多肿瘤细胞都能耐受TRAIL诱导的凋亡[4, 5]。研究已经发现,肝细胞肝癌(hepatocellular carcinoma,HCC)患者对TRAIL治疗存在耐药性,体外观察发现多数肝癌细胞系对TRAIL不敏感[6]。TRAIL与许多化疗药物和蛋白酶等结合使用,可增加肿瘤细胞对TRAIL的敏感度[7],其他药物和TRAIL的联合应用成为肿瘤治疗的研究热点。曲古抑菌素 A(Trichostatin A,TSA)是一种有效的组蛋白去乙酰化酶(histone deacetylase,HDAC)抑制剂,对基因的表观遗传修饰有调节作用,是一种新型的抗肿瘤药物,其能通过抑制蛋白激酶B(protein kinase B,AKT)的活性,诱导卵巢癌细胞的凋亡[8],并能增加前列腺癌细胞对TRAIL的敏感度[9]。然而,TSA是否能促使人肝癌细胞对TRAIL的敏感,目前还不清楚。本研究应用TSA和TRAIL联合处理体外培养的肝癌Bel7402细胞,观察两者抗肿瘤药物对这种肝癌细胞凋亡的影响,并初步探讨药物联合作用的机制,为联合用药治疗肝癌提供新的策略。 1 材料和方法
1.1 主要试剂和仪器
重组人可溶性TRAIL蛋白(PEPROTECH公司),TSA(Sigma公司)、二甲基亚砜(DMSO,Amresco公司)、四甲基偶氮唑盐(3-(4,5-Dimethylthiazol-2-yl)-2,5- diphenyltetrazolium bromide,MTT)(Sigma公司);胎牛血清(Solarbio公司)、胰蛋白酶(Gibco公司),DAPI染液和抗荧光淬灭封片液(上海碧云天生物技术有限公司),p65抗体和H3抗体(Santa Cruze公司),标记FITC的羊抗兔二抗(Abcom公司),标记HRP的羊抗鼠二抗(Santa Cruze公司)。 1.2 细胞培养
人肝癌细胞株 Bel7402(本室保存),细胞培养于高糖DMEM培养液,内含10%胎牛血清、青霉素(100 u/ml)和链霉素(100 u/ml)。培养条件为37℃、5%CO2饱和湿度。 1.3 MTT法检测细胞存活率
取对数生长期细胞,0.25%胰蛋白酶消化,制备细胞悬液。接种细胞于96孔细胞培养板,每孔添加200 μl细胞悬液,细胞数目为1×104个,接种后正常条件(37℃、5%CO2)培养。接种细胞培养24 h后,按照浓度梯度加入TSA(0、10、20、50、100 ng/ml)组培养6、12、24和48 h;而加入TRAIL(0、10、50、100 ng/ml)组则再培养12和24 h。TSA 20 ng/ml预处理4 h后序贯加入不同浓度TRAIL(10、50、100 ng/ml)培养共计24 h。每一浓度不同时间各重复8孔,每板重复3次。药物刺激特定时间后,每孔加入15 μl浓度为5 mg/ml的MTT,37℃、5%CO2饱和湿度条件下孵育,按上述时间孵育后终止培养,弃上清液,每孔加入100 μl DMSO,振荡10 min左右,酶标仪测吸光值(A),波长为490 nm。以未加药的肝癌细胞株Bel7402为对照组,肿瘤细胞存活率(%)=(药物处理组A490/对照组A490)×100%。 1.4 光学显微镜下观察药物处理细胞形态
接种细胞于6孔细胞培养板,待细胞密度80%左右,加药物处理,分别为TSA(20 ng/ml)处理组,TRAIL(100 ng/ml)处理组,TSA(20 ng/ml)和TRAIL(100 ng/ml)联合处理组,处理时间为24 h,以无药物处理组为对照,光学显微镜下观察细胞生长状况,照相留存结果。 1.5 DAPI染色检测细胞凋亡
取生长状态良好的细胞,采用上述方法将细胞接种于6孔板爬片,待细胞密度80%左右,分组和方法1.4相同,以无药物处理组为对照。将药物处理24 h的细胞吸弃培养液后,PBS洗3次,4%的多聚甲醛在室温固定15 min,PBS在室温漂洗2次,每次10 min,用含0.5% Triton-X-100在室温穿孔15 min,PBS在室温漂洗2次,每次10 min,加入DAPI染色液室温作用30 min,此后用锡箔包裹,PBS在室温漂洗2次,每次10 min。在玻片上滴加5 μl抗荧光淬灭剂,将玻片缓慢盖在载玻片上,封片,可放入湿盒中4℃保存。荧光显微镜下观察,用UV波段激发,冷CCD成像,照相保存实验结果。 1.6 免疫细胞化学检测NF-κB/p65表达定位
采用上述方法接种细胞于6孔板内爬片,同样用上述药物处理细胞,以无药物处理组为对照,以TSA(200 ng/ml)为p65蛋白核内转移的阳性参照组。药物处理24 h后,PBS洗3次,每次5 ml,4%多聚甲醛室温固定30 min;取出盖玻片,PBS室温洗3次,5% BSA封闭1.5 h;PBS室温洗3次,0.1% Triton-X-100室温穿孔15 min后完全吸除,加入PBS稀释的NF-κB/p65抗体,4℃孵育过夜;吸掉抗体,PBS洗3次;以下避光操作,加入稀释的荧光二抗,37℃孵育1 h;吸除二抗,PBS洗3次,加入DAPI染细胞核,室温15 min;吸除DAPI染液,PBS室温漂洗3次,载玻片中央滴加抗荧光衰减封片剂,将盖玻片盖在载玻片上,封片,荧光倒置显微镜观察,用UV波段激发,观察DAPI染的细胞核;490 nm激发,观察FITC标记的绿色荧光,冷CCD成像,照相保存实验结果。 1.7 Western blot检测细胞核蛋白中p65蛋白的表达
应用核蛋白提取试剂盒[天根生化科技(北京)有限公司]提取不同药物处理组细胞核蛋白,Bradford法定量,取60 μg蛋白上样,10% SDS-PAGE胶电泳,40 V转膜3 h,PBS洗膜5 min,洗3次,5%脱脂奶粉封闭2 h,PBST洗膜3次,每次10 min,p65或H3一抗4℃过夜孵育,同样方法洗膜,二抗室温孵育2 h,PBST洗膜3次,ECL发光仪成像,应用软件进行灰度值扫描。 1.8 统计学方法
采用SPSS10.0软件进行统计学处理。实验重复3次。用方差分析和配对t检验。 P<0.05为差异有统计学意义。 2 结果
2.1 TSA对肝癌细胞Bel7402的毒性作用
不同浓度(0、10、20、50和100 ng/ml)TSA处理人肝癌Bel7402细胞系6、12、24和48 h,用MTT法检测细胞生长,结果显示,不同浓度的TSA单独处理的各个时间点对Bel7402细胞生长没有明显抑制作用,与对照组(0 ng/ml)比较差异无统计学意义(P>0.05);但是,TSA作用48 h后,对肝癌细胞Bel7402的增殖有明显抑制,不同浓度(10、20、50和100 ng/ml)处理细胞存活率分别为(90.84±5.4)%、(86.62±12.3)%、(61.38±11.1)%、(48.75±4.4)%;50和100 ng/ml TSA处理的细胞存活率与对照组比较差异有统计学意义(P=0.036和P=0.021),见图 1。TSA浓度≤20 ng/ml时,细胞生长抑制率几乎不受作用时间影响,因此,在以下的实验中选择20 ng/ml TSA为联合用药的浓度。
|
| *: P<0.05, compared with control group (0ng/ml) 图 1 不同浓度TSA作用不同时间对Bel7402细胞增殖的影响 (n=8) Figure 1 Effects of different concentrations of TSA on proliferation of Bel7402 cells at different time (n=8) |
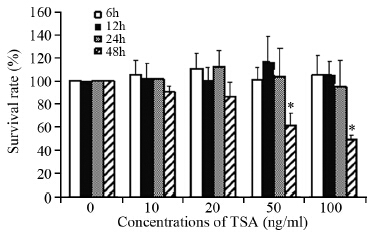
不同浓度(0、10、50和100 ng/ml)的TRAIL分别处理人肝癌Bel7402细胞12和24 h后,MTT检测结果显示,12 h时Bel7402细胞增殖无抑制;而24 h时,不同浓度(10、50和100 ng/ml)的TRAIL分别处理人肝癌Bel7402细胞的存活率分别为(99.7±3.1)%、(93.6±1.23)%、(84.9±5.0)%,提示,大剂量TRAIL对细胞的生长有一定的抑制作用,但差异无统计学意义(P>0.05),见图 2。由于TRAIL浓度在100 ng/ml时,肿瘤细胞抑制率<20%,因此,在以下的实验中选择100 ng/ml 的TRAIL为联合用药的浓度。
|
| 图 2 不同浓度的TRAIL处理12和24h对Bel7402细胞增殖的影响 (n=8) Figure 2 Effects of different concentrations of TRAIL on proliferation of Bel7402 cells for 12 and 24h (n=8) |
浓度为20 ng/ml的TSA预处理4 h后,分别给予10、50、100 ng/ml的TRAIL处理细胞24 h,MTT法检测Bel7402细胞存活率。结果显示,随着TRAIL浓度增加,细胞存活率逐渐降低,分别为(85.7±4.7)%、(69.8±9.2)%、(57.1±5.4)%,呈剂量依赖性。20 ng/ml的TSA预处理与100 ng/ml的TRAIL共作用24 h细胞抑制率最高,抑制率>40%,与对照组比较差异有统计学意义(P=0.021)。提示,TSA能增加Bel7402细胞对TRAIL的敏感度。 2.4 TSA促进TRAIL诱导肝癌Bel7402细胞的凋亡
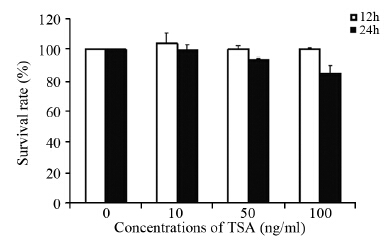
用20 ng/ml TSA、100 ng/ml TRAIL以及20 ng/ml TSA预处理4 h后再用100 ng/ml TRAIL共同作用,经处理24 h后,光学显微镜下观察细胞形态和数目的改变。结果显示,单独用药处理组与对照组相比没有明显形态上的改变,见图 3A~C,而药物联合处理组细胞数明显减少,多数细胞变形、体积变小,并出现皱缩、变圆、脱落现象,见图 3D。
|
| A-D: ×100; E-H: ×400; White arrows indicated cells apoptosome. The images were the representation of three independent experiments 图 3 20ng/ml TSA联合100ng/ml TRAIL诱导肝癌细胞Bel7402的凋亡的作用 Figure 3 TSA(20ng/ml) combined with TRAIL(100ng/ml) induced apoptosis of Bel7402 cells |
为了证明经药物处理后细胞发生凋亡,我们对上述各组细胞进行DAPI染色,用荧光显微镜观察细胞核成蓝色荧光,单独用药处理组与对照组相比,细胞核形态未见明显异常,见图 3E~G。TSA(20 ng/ml)预处理后再联合TRAIL(100 ng/ml)处理,结果显示,多数细胞的染色质浓缩,核膜裂解、染色质分割成块状和凋亡小体等典型的凋亡形态出现,见图 3H。 2.5 TSA和TRAIL联合处理对Bel7402细胞的NF-κB/p65定位和表达的影响
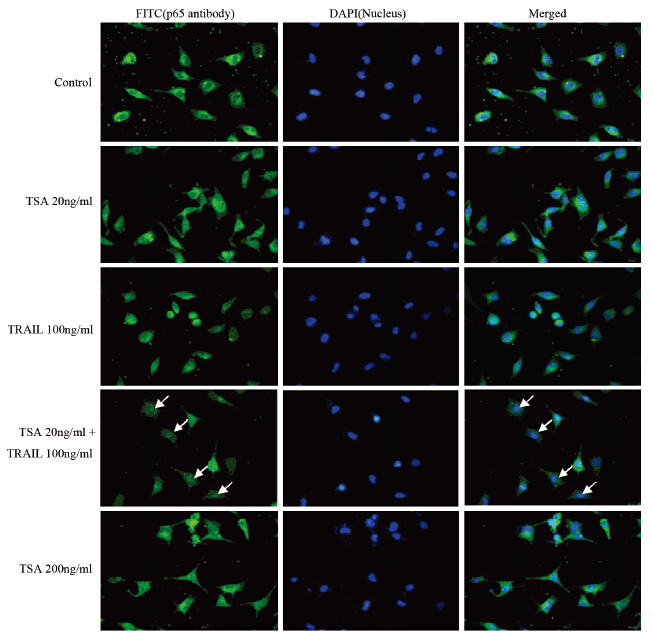
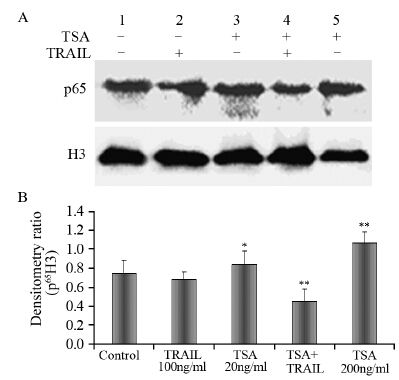
TSA(20 ng/ml)、 TRAIL(100 ng/ml)单独处理以及TSA预处理4 h再用TRAIL处理细胞24 h后,荧光显微镜下观察FITC标记的绿色荧光、DAPI染色的细胞核蓝色荧光,并通过软件将两者合并。荧光显微镜观察显示,对照组细胞p65蛋白(绿色荧光)在细胞质和细胞核均有分布,但主要分布在胞质中;用小剂量TSA(20 ng/ml)处理的细胞,p65蛋白有微量核内转移,而大剂量TSA(200 ng/ml)处理的细胞,与对照组和TSA(20 ng/ml)处理组细胞比较,p65蛋白(绿色荧光)发生明显核内转移,趋于全细胞均匀分布;两种药物联合处理的细胞,p65蛋白(绿色荧光)显著减弱,胞质中绿色荧光发生明显核内转移,见图 4。应用Western blot方法检测细胞核蛋白内p65表达,结果显示,两种药物联合作用的细胞核蛋白p65的表达明显低于其他药物处理组,大剂量TSA(200 ng/ml)处理组的细胞p65蛋白在核内表达高于其他药物处理组。统计学分析显示,单独TSA(20 ng/ml)处理也能增加p65表达,和对照组、单独TRAIL处理组比较,差异有统计学意义(P=0.043);而TSA(20 ng/ml)和TRAIL(100 ng/ml)联合处理,能显著性降低细胞p65表达,和对照组、TSA(20 ng/ml)组、TRAIL组比较差异均有统计学意义(P<0.01),见图 5A、B。
|
| White arrows indicated the nucleus translocation of p65 图 4 荧光显微镜观察免疫细胞化学检测后NF-κB/p65在亚细胞中的定位 (×400) Figure 4 Subcellular location of NF-κB/p65 investigated by immunocytochemistry under a inverted fluorescence microscope (×400) |
|
| A: NF-κB/p65 protein expression in cell nucleuses in different treatment groups investigated by Western blot; 1: Control group;2: TRAIL(100ng/ml)treatment group; 3: TSA(20ng/ml)treatment group; 4: TSA(20ng/ml)+ TRAIL(100ng/ml)treatment group; 5: TSA(200ng/ml) treatment group; H3: histone 3, as internal control; B: densitometry ratio of protein expression and internal control H3; *: P<0.05, compared with control group and TRAIL(100ng/ml)treatment group; **: P<0.01,compared with control group, TRAIL(100ng/ml) treatment group and TSA (20ng/ml)treatment group 图 5 Western blot分析不同药物处理后细胞核中NF-κB/p65蛋白的表达 (n=3) Figure 5 NF-κB/p65 protein expression in cell nucleuses in different treatment groups investigated by Western blot (n=3) |
TSA是HDAC抑制剂的代表药物之一,特异抑制HDAC的活性,促使组蛋白及非组蛋白的赖氨酸残基持续乙酰化,改变染色体结构和蛋白质的功能,导致肿瘤细胞的形态和功能发生改变,从而发挥抑制细胞增殖、诱导肿瘤细胞分化和(或)凋亡等作用,是具有较好应用前景的抗肿瘤药物之一[10, 11]。本研究单独应用TSA(≤50 ng/ml)处理肝癌Bel7402细胞6、12和24 h,细胞增殖抑制没有随药物浓度和作用时间改变而发生明显变化;而处理细胞48 h,细胞增殖抑制率随药物浓度升高而增高,表明TSA对于肝癌Bel7402细胞的生长抑制作用具有时间依赖性。在其他肝癌细胞系的研究中表明,TSA能明显抑制肿瘤细胞的增殖,并促进凋亡,其作用具有时间和剂量依赖性[12, 13],在处理48 h后作用更加明显[14]。虽然与本研究中细胞系存在差异,但是TSA的作用模式和剂量都存在相似性。本研究还发现正常肝细胞系L-02在TSA浓度为50 ng/ml处理24 h即对L-02有明显抑制作用,其对TSA的耐受低于肝癌Bel7402细胞,TSA抑制L-02细胞增殖的时间和剂量依赖性更加明显(未公布的结果)。也就是TSA对正常肝细胞也有一定的毒性作用,但是本研究在体外开展,不能说明体内复杂的药物代谢后是否对正常肝细胞的产生毒性效应。本研究在联合药物作用时,选取TSA对肝癌Bel7402细胞无作用的药物浓度和时间进行,利于观察药物的协同作用,结果显示,TSA能增加肝癌Bel7402细胞对TRAIL的敏感度。
TRAIL作为一种有效的基因治疗药物具有广泛的应用前景,其参与机体的多种免疫过程,对病毒感染、肿瘤和自身免疫缺陷病等都有一定的抑制作用。其能与死亡受体结合,启动凋亡信号转导途径,诱导肿瘤细胞凋亡,而对正常组织和细胞没有明显的毒副作用[15, 16]。TRAIL在体内的确切作用和抑癌机制尚不十分清楚,尤其是对肿瘤细胞与正常细胞选择性凋亡的作用机制和耐受机制等还需要阐明。多种肝癌细胞系对TRAIL都不敏感,而与化疗、放疗、药物蛋白酶体抑制因子等相结合使用,可增加肿瘤细胞对TRAIL的敏感度,进而提高对肿瘤细胞的杀灭疗效[17],因此,TRAIL与化疗药物协同作用机制和安全性研究有利于其在临床中的应用。本研究以肝癌细胞系Bel7402为对象,TRAIL单独处理12 h,对细胞的增殖没有明显的影响,而处理24 h,细胞增殖受到轻度抑制,但差异无统计学意义。因此,本研究应用TSA预处理后加入TRAIL共同作用24 h,细胞增殖明显受到抑制(抑制率大于40%),并出现大量细胞凋亡。
NF-κB是一种能与免疫球蛋白κ轻链基因κB序列特异结合并调节其转录的核蛋白因子。在哺乳动物细胞中NF-κB家族由RelA(p65)、c-Rel、RelB、NF-κB1(p105)和NF-κB2(p100)组成。非激活状态下,NF-κB以无活性的形式存在于胞质中[18],受到刺激后,NF-κB活化转位入核,活化的NF-κB以二聚体的形式存在,异源二聚体p50/RelA具有转录激活活性,而同源二聚体p50/p50或p52/p52具有转录抑制活性[19]。TRAIL信号通路中NF-κB的作用存在争议,一部分研究认为NF-κB的活化导致细胞对TRAIL诱导的细胞凋亡不敏感[20, 21],而另一部分研究则认为NF-κB的活化促进TRAIL诱导的细胞凋亡[22, 23],TRAIL通过抑制NF-κB依赖的cFLIP表达诱导肿瘤细胞凋亡[24, 25],Chen等[23]认为,产生这种相反结果可能是由于NF-κB不同亚基功能和表达量的差异导致。该研究认为,RelA(p65)亚基过表达抑制肿瘤凋亡,而c-Rel过表达则促进细胞凋亡。近期,Zhang等[26]研究发现TRAIL通过抑制NF-κB亚基IkBα的磷酸化和p65的表达以及核转移,诱导肿瘤细胞凋亡。本研究应用免疫细胞化学技术,研究NF-κB的p65亚基在细胞中的表达定位,结果显示,单独应用剂量为20 ng/mlTSA处理细胞,p65亚基主要存在于细胞质,而200 ng/mlTSA处理细胞,发现胞质中p65明显向核转移,说明TSA活化NF-κB并发生核转位;单独应用100 ng/mlTRAIL处理细胞,同样发现NF-κB的核转移;而两种药物联合应用后,NF-κB/p65发生明显核转移现象,积聚在核内一点,并且绿色荧光明显减弱,可能其表达降低。本研究通过RT-PCR证实其表达下降(未公布结果),但是降低并不显著;Western blot结果同样证实,两种药物联合应用后,细胞核内p65蛋白明显低于其他药物组。其他研究表明,10 ng/mlTSA能活化NF-κB/p65,但却抑制其与DNA的结合活性,具体机制还不清楚。本研究结果提示TSA预处理联合TRAIL促进细胞凋亡可能与RelA(p65)表达下降和DNA结合活性降低相关,但RelA(p65)核移位后如何发挥其诱导肿瘤细胞凋亡还值得进一步探索。
本研究结果认为,TSA和TRAIL联合作用,能提升人肝癌Bel7402细胞对TRAIL的敏感度,是一种有效的诱导肝癌细胞凋亡的药物组合模式。
| [1] | Wiley SR, Schooley K, PJ Smolak, et al. Identification and characterization of a new member of the TNF family that induces apoptosis[J]. Immunity, 1995, 3(6): 673-82. |
| [2] | Gura T. How TRAIL kills cancer cells, but not normal cells[J]. Science, 1997, 277(5327): 768. |
| [3] | Fiorucci G, Vannucchi S, Chiantore MV, et al. TNF-related apoptosis-inducing ligand(TRAIL) as a pro-apoptotic signal transducer with cancer therapeutic potential[J]. Curr Pharm Des, 2005, 11(7): 933-44. |
| [4] | Menke C, Bin L, Thorburn J, et al. Distinct TRAIL resistance mechanisms can be overcome by proteasome inhibition but not generally by synergizing agents[J]. Cancer Res, 2011, 71(5): 1883-92. |
| [5] | Belkhiri A, Zhu S, Chen Z, et al. Resistance to TRAIL is mediated by DARPP-32 in gastric cancer[J]. Clin Cancer Res, 2012, 18(14): 3889-900. |
| [6] | Song IS, Jun SY, Na HJ, et al. Inhibition of MKK7-JNK by the TOR signaling pathway regulator-like protein contributes to resistance of HCC cells to TRAIL-induced apoptosis[J]. Gastroenterology, 2012, 143(5): 1341-51. |
| [7] | Tomek S, Horak P, Pribill I, et al. Resistance to TRAIL-induced apoptosis in ovarian cancer cell lines is overcome by co-treatment with cytotoxic drugs[J]. Gynecol Oncol, 2004, 94(1): 107-14. |
| [8] | Zhou C, Qiu L, Sun Y, et al. Inhibition of EGFR/PI3K/AKT cell survival pathway promotes TSA’s effect on cell death and migration in human ovarian cancer cells[J]. Int J Oncol, 2006, 29(1): 269-78. |
| [9] | Taghiyev AF, Guseva NV, Sturm MT, et al. Trichostatin A(TSA) sensitizes the human prostatic cancer cell line DU145 to death receptor ligands treatment[J]. Cancer Biol Ther, 2005, 4(4): 382-90. |
| [10] | Armeanu S1, Pathil A, Venturelli S, et al. Apoptosis on hepatoma cells but not on primary hepatocytes by histone deacetylase inhibitors valproate and ITF2357[J]. J Hepatol, 2005, 42(2): 210-7. |
| [11] | Jasek E, Lis GJ, Jasinska M, et al. Effect of histone deacetylase inhibitors trichostatin A and valproic acid on etoposide-induced apoptosis in leukemia cells[J]. Anticancer Res, 2012, 32(7): 2791-9. |
| [12] | Nakamura M, Saito H, Ebinuma H, et al. Reduction of telomerase activity in human liver cancer cells by a histone deacetylase inhibitor[J]. J Cell Physiol, 2001, 187(3): 392-401. |
| [13] | Carlisi D, Vassallo B, Lauricella M, et al. Histone deacetylase inhibitors induce in human hepatoma HepG2 cells acetylation of p53 and histones in correlation with apoptotic effects[J]. Int J Oncol, 2008, 32(1): 177-84. |
| [14] | Ganslmayer M, Konturek P, Herold C, et al. Antitumoral efficacy of four histone deacetylase inhibitors in hepatoma in vitro and in vivo[J]. Anticancer Res, 2012, 32(12): 5263-9. |
| [15] | Pati?o-García A, Zalacain M, Folio C, et al. Profiling of chemonaive osteosarcoma and paired-normal cells identifies EBF2 as a mediator of osteoprotegerin inhibition to tumor necrosis factor-related apoptosis-inducing ligand-induced apoptosis[J]. Clin Cancer Res, 2009, 15(16): 5082-91. |
| [16] | Mucha SR, Rizzani A, Gerbes AL, et al. JNK inhibition sensitises hepatocellular carcinoma cells but not normal hepatocytes to the TNF-related apoptosis-inducing ligand[J]. Gut, 2009, 58(5): 688-98. |
| [17] | Yamanaka T, Shiraki K, Sugimoto K, et al. Chemotherapeutic agents augment TRAIL-induced apoptosis in human hepatocellular carcinoma cell lines[J]. Hepatology, 2000, 32(3): 482-90. |
| [18] | Chen LF, Greene WC. Shaping the nuclear action of NF-kappaB[J]. Nat Rev Mol Cell Biol, 2004, 5(5): 392-401. |
| [19] | Motoyama M, Yamazaki S, Eto-Kimura A, et al. Positive and negative regulation of nuclear factor-kappaB-mediated transcription by IkappaB-zeta, an inducible nuclear protein[J]. J Biol Chem, 2005, 280(9): 7444-51. |
| [20] | Kim YS, Schwabe RF, Qian T, et al. TRAIL-mediated apoptosis requires NF-kappaB inhibition and the mitochondrial permeability transition in human hepatoma cells[J]. Hepatology, 2002, 36(6): 1498-508. |
| [21] | Ehrhardt H, Fulda S, Schmid I, et al. TRAIL induced survival and proliferation in cancer cells resistant towards TRAIL-induced apoptosis mediated by NF-kappaB[J]. Oncogene, 2003, 22(25): 3842-52. |
| [22] | Shetty S, Gladden JB, Henson ES, et al. Tumor necrosis factor-related apoptosis inducing ligand(TRAIL) up-regulates death receptor 5 (DR5) mediated by NFkappaB activation in epithelial derived cell lines[J]. Apoptosis, 2002, 7(5): 413-20. |
| [23] | Chen X, Kandasamy K, Srivastava RK. Differential roles of RelA (p65) and c-Rel subunits of nuclear factor kappa B in tumor necrosis factor-related apoptosis-inducing ligand signaling[J]. Cancer Res, 2003, 63(5): 1059-66. |
| [24] | Seo OW, Kim JH, Lee KS, et al. Kurarinone promotes TRAIL-induced apoptosis by inhibiting NF-κB-dependent cFLIP expression in HeLa cells[J]. Exp Mol Med, 2012, 44(11): 653-64. |
| [25] | Luo JL, Maeda S, Hsu LC, et al. Inhibition of NF-kappaB in cancer cells converts inflammation- induced tumor growth mediated by TNFalpha to TRAIL-mediated tumor regression[J]. Cancer Cell, 2004, 6(3): 297-305. |
| [26] | Zhang JS, Herreros-Vilanueva M, Koenig A, et al. Differential activity of GSK-3 isoforms regulates NF-κB and TRAIL or TNFα induced apoptosis in pancreatic cancer cells[J]. Cell Death Dis, 2014, 5: e1142. |
 2015, Vol. 42
2015, Vol. 42