文章信息
- 杨青青,邓燕飞. 2015.
- YANG Qingqing, DENG Yanfei. 2015.
- 特异长链非编码RNA在鼻咽癌中的表达及其意义
- Expression of Specific Long Non-coding RNA in Nasopharyngeal Carcinoma and Related Significance
- 肿瘤防治研究, 2015, 42(03): 219-224
- Cancer Research on Prevention and Treatment, 2015, 42(03): 219-224
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2015.03.002
-
文章历史
- 收稿日期:2014-03-28
- 修回日期:2014-08-11
2. 361004 厦门,厦门大学附属中山医院耳鼻咽喉科
2. Department of Otolaryngology, Zhongshan Hospital, Xiamen University, Xiamen 361004, China
鼻咽癌是中国南方及东南亚地区发病率较高 的一种恶性肿瘤,其在广东省的发病率大约是欧 洲和北美的100倍[1]。鼻咽癌起病隐匿、早期诊断 困难,大多数患者就诊时已达中晚期。近年来, 尽管鼻咽癌的诊治水平有了较大的提高,但鼻咽 癌患者的5年生存率仍只有70%左右[2]。目前,鼻 咽癌发病的分子机制尚不很清楚,因此,深入研究鼻咽癌的发生发展机制有利于提高鼻咽癌的防 治水平。
越来越多的研究表明,长链非编码RNA(long non-coding RNA,lncRNA)与人类多种肿瘤密切 相关。全基因组关联分析发现lncRNA可能通过表 观遗传调控、转录调控和转录后调控等机制[3]来 参与肿瘤的发生和发展。研究发现,lncRNA在胶 质瘤、肝癌、胰腺癌等多种恶性肿瘤中的表达水 平明显改变,提示lncRNA可能是潜在的致癌或抑 癌基因[4, 5]。目前,鼻咽癌中lncRNA的研究报道较 少,且主要是单个lncRNA的研究[6, 7],其在鼻咽癌 发生发展中的作用亟待进一步研究。
本实验通过基因芯片技术全基因组水平检测 lncRNA在鼻咽癌与慢性鼻咽炎组织中的表达水 平,运用生物信息学技术预测lncRNA在鼻咽癌中 的潜在功能,阐明其在鼻咽癌发生发展中可能的 分子调控机制,为寻找诊治鼻咽癌新型特异分子 标志物提供实验基础。 1 资料与方法 1.1 资料
收集厦门大学附属中山医院耳鼻咽喉科2013 年8月—2013年10月经病理确诊的12例活检组织标 本,其中鼻咽癌6例、慢性鼻咽炎6例,置于-80℃ 冰箱保存,所有患者活检前均未接受过放化疗。 1.2 实验方法 1.2.1 总RNA抽提与质量检测
按照TRIzol试剂 盒(Invitrogen公司)说明书,分别提取6例鼻咽癌 和6例慢性鼻咽炎组织标本RNA,使用NanoDrop ND-1000,测定样品RNA在分光光度计260、280、 230 nm的吸光度值,计算RNA浓度并评估纯度。 本实验要求总RNA的吸光度OD260/OD280比值介于 1.8~2.1,越接近2.0说明纯度越高。OD260/OD280的 比值<1.8或>2.1,都说明RNA被污染。然后使用 甲醛变性琼脂糖凝胶电泳检测RNA纯度及完整 性,根据5S、18S、28S条带的宽度及亮度判断。 1.2.2 cDNA样品合成、标记和杂交
利用 Invitrogen公司提供的ds-cDNA合成试剂盒 (Quick Amp Labeling Kit)进行反转录,合成双 链互补DNA;利用Agilent公司提供的单色DNA 标记试剂盒进行双链互补DNA Cy3荧光标记,使 用NanoDrop ND-1000分光光度计检测荧光标记 效率,对荧光信号强度精确测定是实现检测特异 性的基础,可以确保后续芯片杂交实验结果的准 确性和可靠性;利用Agilent公司提供的Gene Expression Hybridization Kit进行芯片杂交,所用芯 片为美国Arraystar Human LncRNA Microarray V3.0(8×60K),可以检测30 586个lncRNA和 26 109个mRNA。然后利用Agilent公司提供的 基因表达清洗缓冲液1和2进行清洗。 1.2.3 图像采集和数据分析
利用Agilent公司 提供的Agilent Microarray Scanner G2505C芯片扫 描仪进行基因芯片扫描,所得各标本Cy3荧光扫 描灰度图输入Agilent Feature Extraction Software (version 11.0.1.1)图像处理软件进行数据分 析,用分位数和强多芯片平均法对基因表达数据 进行归一化。所有lncRNA数据由GeneSpring GX v11.5.1 software package(Agilent Technologies) 进行分析。利用火山图(Volcano plot)筛选2倍 以上差异表达的lncRNA。然后按lncRNA表达水 平进行聚类分析。GO分析和Pathway分析用于分 析差异表达的mRNA。Pathway分析所用数据库为 KEGG 数据库(Kyoto Encyclopedia of Genes and Genomes database)。 1.3 统计学方法
应用SPSS19.0统计软件进行Student’s t检验和 Fisher精确检验,P<0.05为差异有统计学意义。 2 结果 2.1 总RNA质量分析
NanoDrop ND-1000分光光度计测定12份样品 的总RNA的OD260/OD280比值均在1.8~2.1之间。总 RNA经甲醛变性琼脂糖凝胶电泳共显示出3条条带, 见图 1,说明总RNA质量及纯度好,可用于进一步 的实验分析。

|
| 1-12: number of 12 samples 图 1 12例鼻咽癌及慢性鼻咽炎样品电泳图谱 Figure 1 Electrophoretogram of 12 nasopharyngeal carcinoma and chronic nasopharyngitis samples |
利用芯片比较分析鼻咽癌组与慢性鼻咽炎组 样品的lncRNA表达谱,其中变化在2倍以上且 差异具有统计学意义(P<0.05)的lncRNA认为 是差异表达的lncRNA,共856条,占总lncRNA 2.80%(856/30 586);其中上调的共425条,下 调的共431条;5倍以上变化的共39条,其中上调的共27条,下调的共12条;10倍以上变化的共9 条,其中上调的共8条,下调的共1条;20倍以上 变化的共3条,均上调;30倍以上变化的共2条, 均上调,见表 1。
 |
利用火山图筛选差异有统计学意义 (P<0.05)并且变化倍数在2.0倍以上的lncRNA。 火山图中所有红点代表鼻咽癌与慢性鼻咽炎组 织中差异表达的lncRNA(P<0.05,fold change﹥ 2.0),见图 2。散点图可直观的观察差异表达的 lncRNA在鼻咽癌组与慢性鼻咽炎组之间的相关 性。散点图X轴和Y轴的值都是标准化处理后的信 号值。绿色的线表示折叠倍率线,折叠倍率≥2, 见图 3。差异表达的lncRNA分别在鼻咽癌与慢性鼻 咽炎组的表达水平变量值呈正相关关联。

|
| Red: differentially expressed lncRNA with more than two-fold change (P<0.05) 图 2 鼻咽癌与慢性鼻咽炎组织中lncRNA变化的火山图 Figure 2 Volcano plot of lncRNA variation between nasopharyngeal carcinoma and chronic nasopharyngitis tissues |

|
| Green line: fold change line 图 3 鼻咽癌与慢性鼻咽炎组织中lncRNA变化的散点图 Figure 3 Scatter plot of lncRNA variation between nasopharyngeal carcinoma and chronic nasopharyngitis tissues |
利用聚类分析方法分析所有的l n cRNA, 并按照lncRNA的表达水平将其分成3类:增强 子型lncRNA、HOX基因簇、基因间lncRNA。 分别是1 569个增强子型lncRNAs,95个HOX基 因簇(蛋白编码基因40个,非蛋白编码基因55 个),10 540个基因间lncRNAs,并定位邻近的 编码基因。生物信息学技术分析发现lncRNA表 达水平与邻近mRNA表达有关,lncRNA可能通 过与邻近编码转录本或蛋白质相互作用而参与 鼻咽癌的发生发展,见图 4。

|
| Column: sample; Row: lncRNA; Red: up-regulated lncRNA expression; Green: down-regulated lncRNA expression 图 4 12例样本中856条差异表达lncRNAs的聚类分析热图 Figure 4 Clustering heat map of 856 differentially expressed lncRNAs in 12 samples |
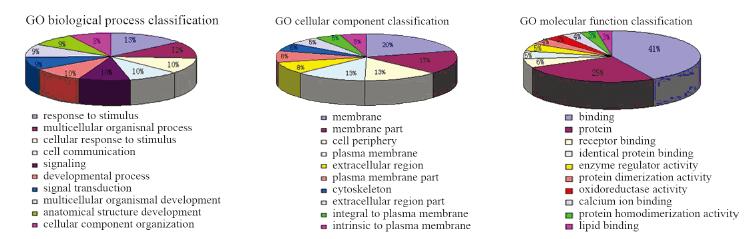
GO分析将mRNA分成3类:生物学过程类 (biological process)、细胞组分类(cellular component) 、分子功能类(molecular function)。表 达上调的3类mRNA的构成比,见图 5。
|
| 图 5 GO富集分析结果 Figure 5 GO enrichment analysis results |
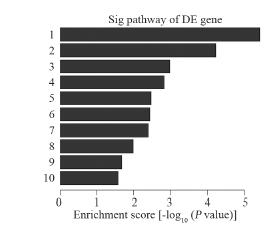
实验结果显示lncRNA潜在的靶基因mRNA 可能参与的信号通路共有2 8 条,其中表达上 调的信号通路18条,下调的13条,有3条信号 通路(Cytokine-cytokine receptor interaction, Chemokine signaling pathway,Neuroactive ligandreceptor interaction)同时参与了表达上调和下调。 上调mRNA富集度较高的10条信号通路中,细胞 因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction)信号通路的富集度最高,为 5.428282,见图 6。
|
| 1: Cytokine-cytokine receptor interaction - Homo sapiens(human); 2: Influenza A - Homo sapiens(human); 3: Measles - Homo sapiens(human);4:Glycine,serine and threonine metabolism - Homo sapiens(human); 5: Jak-STAT signaling pathway - Homo sapiens(human); 6: Hepatitis C-Homo sapiens(human); 7: Herpes simplex infection - Homo sapiens(human); 8: Chagas disease (American trypanosomiasis) - Homo sapiens(human); 9: Type I diabetes mellitus - Homo sapiens(human); 10: Malaria - Homo sapiens(human) 图 6 差异表达上调的mRNA富集度较高的10条信号通路 Figure 6 Signaling pathways of up-regulated mRNA with top ten enrichment score [-log10 (P value)] |
lncRNA是指长度超过200个核苷酸、具有调 控基因表达作用的非编码RNA。众多研究证明 lncRNA具有重要的生物学功能,例如:参与X染 色体沉默、基因组印迹、转录调控和转录后调控 等[8, 9]。lncRNA与相关的编码基因相互作用可以 调节该编码基因关联的蛋白所参与的信号通路, lncRNA的表达水平发生变化,可能导致相应疾病 的发生。近年来,随着lncRNA在多种疾病尤其是 癌症的发生发展中的作用逐渐被揭示,lncRNA的 分子调控机制已经成为人类遗传学研究的一大热 点。迄今为止,鼻咽癌有关lncRNA的研究报道较 少[6, 7, 10],且主要为少数lncRNA的表达研究,lncRNA 在鼻咽癌全基因组水平的表达谱分析及其分 子调控机制仍未见详细阐述。
本实验采用的美国Arraystar lncRNA V3.0芯片覆盖目前所有权威数据库所注释的lncRNA。 在本实验中,我们利用基因芯片技术在全基因组 水平研究了lncRNA、mRNA在鼻咽癌与慢性鼻 咽炎组织中的表达谱,对差异表达的lncRNA、 mRNA进行了分类,并通过生物信息学技术进行 进一步分析。结果显示:差异表达的lncRNA共 856条,其中上调的共425条,下调的共431条;10 倍以上变化的共9条,其中上调的共8条,下调的 共1条。差异表达的mRNA共767条,其中上调的 共426条,下调的共341条。而Gao等[10]利用相关 lncRNA数据库(Microarray data set GSE12452)进 行lncRNA筛选,得出5个在鼻咽癌与正常鼻咽上 皮组织中差异表达的lncRNA,即lnc-C22orf32-1、 lnc-TLR4-1、lnc-BCL2L11-3、lnc-AL355149.1-1、 lnc-ZNF674-1,远少于本实验结果(856条), 可能是因为我们所使用的芯片所覆盖的数据库较 多。我们推测差异表达的lncRNA可能与鼻咽癌的 发生发展有关,lncRNA变化倍数越高,其与鼻咽癌 发生发展的关系可能越密切。根据lncRNA的上调和 下调可将lncRNA分成促癌lncRNA和抑癌lncRNA。 以往的研究证明lncRNA Xist具有参与X染色体失 活、基因印迹和染色质修饰等功能[8],本实验结果 显示差异表达在10倍以上的lncRNA共9条,其中有 5条均位于X染色体,分别是ENST00000417942、 ENST00000434839、uc004ebm.1、ENST00000416330、 ENST00000421322,这些lncRNA是否与X染色体的失 活有关还有待进一步研究。
我们运用聚类分析方法按表达水平将lncRNA 进行分类,并定位邻近的编码基因。生物信息学 技术分析发现lncRNA表达水平与邻近mRNA表 达有关,lncRNA可能通过与邻近编码转录本或 蛋白质相互作用而参与鼻咽癌的发生发展。因 此,我们推测lncRNA邻近的编码基因可能是相 应lncRNA的潜在靶基因,lncRNA可能通过调节 邻近编码基因的表达水平以促进或抑制鼻咽癌的 发生。本实验主要根据芯片筛选的差异表达的 lncRNA及lncRNA邻近编码基因(位于lncRNA 转录本两侧<300 kb)数据结果来分析,其中5个 lncRNA,即uc001yfd.1、ENST00000558147、 ENST0 0 0 0 0 5 3 7 1 9 2、ENST0 0 0 0 0 5 6 9 6 5 5、 uc003ihb.3,邻近的编码基因分别为C14orf132、 SPESP1/NOX5、NTF3、SSTR2、PCDH10。GO 分析将mRNA分成3类,并分析了mRNA在细胞 生物学方面的主要功能,结果显示分子功能类 上调的mRNA中参与结合功能的mRNA所占的比 例最大,大约为42%。Pathway分析显示mRNA 共参与28条信号通路,其中参与细胞因子-细胞 因子受体相互作用[Cytokine-cytokine receptor interaction-Homo sapiens(human)]信号通路的 上调差异表达的mRNAs的富集度最高,说明这 条信号通路可能在鼻咽癌的发生发展中发挥重要 作用。每个lncRNA可能有多个靶基因,每个靶 基因的mRNA又可能受到多个lncRNA的调控, 从而构成复杂的多基因调控网络。因此,根据本 实验Pathway分析差异表达的mRNA所参与的信 号通路,今后可选取一些mRNA作为靶基因进行 编码-非编码(coding-non-coding,CNC)基因 分析,找出lncRNA的靶基因mRNA,绘制CNC (coding-non-coding)网络图,确认与鼻咽癌相 关的lncRNA作为特异分子标志物,为鼻咽癌的 筛查和诊治提供新思路。(芯片实验由上海康成 生物工程有限公司完成,特此致谢!)
| [1] | Yu MC, Yuan JM. Epidemiology of nasopharyngeal carcinoma[J].Semin Cancer Biol, 2002, 12(6): 421-9. |
| [2] | Wu F, Wang R, Lu H, et al. Concurrent chemoradiotherapy inlocoregionally advanced nasopharyngeal carcinoma: Treatmentoutcomes of a prospective, multicentric clinical study[J].Radiother Oncol, 2014, 112(1): 106-11. |
| [3] | Ponting CP, Oliver PL, Reik W. Evolution and functions of longnoncoding RNAs[J]. Cell, 2009, 136(4): 629-41. |
| [4] | Maruyama R, Suzuki H. Long noncoding RNA involvement incancer[J]. BMB Rep, 2012, 45 (11): 604-11. |
| [5] | Huarte M, Rinn JL. Large non-coding RNAs: missing links incancer[J]. Hum Mol Genet, 2010, 19(R2): R152-61. |
| [6] | Nie Y, Liu X, Qu S, et al. Long non-coding RNA HOTAIR is anindependent prognostic marker for nasopharyngeal carcinomaprogression and survival[J]. Cancer Sci, 2013, 104(4): 458-64. |
| [7] | Xie LY, Hu ZY, Wang XY, et al. Expression of long noncodingRNA MALAT1 gene in human nasopharyngeal carcinoma celllines and its biological significance[J]. Nan Fang Yi Ke Da XueXue Bao, 2013, 33(5): 692-7. [谢林英, 胡志燕, 王晓燕, 等. 长非编码MALAT1基因在人鼻咽癌细胞株的表达及生物学意义[J].南方医科大学学报, 2013, 33(5): 692-7.] |
| [8] | Lee JT. Lessons from X-chromosome inactivation: long ncRNA asguides and tethers to the epigenome[J]. Genes Dev, 2009, 23(16):1831-42. |
| [9] | Gibb EA, Brown CJ, Lam WL. The functional role of long noncodingRNA in human carcinomas[J]. Mol Cancer, 2011, 10: 38. |
| [10] | Gao W, Chan JYW, Wong TS. Differential expression of longnoncoding RNA in primary and recurrent nasopharyngealcarcinoma[J]. Biomed Res Int, 2014, 2014: 404567. |
 2015, Vol. 42
2015, Vol. 42


