文章信息
- 张建波,吕晓东,宋魏,于庆凯,刘明阁,胡骏,房百俊.
- ZHANG Jianbo, LV Xiaodong, SONG Wei, YU Qingkai, LIU Mingge, HU Jun, FANG Baijun.
- 乳腺癌淋巴结微转移患者克隆性T细胞TCRα链CDR3谱型分析
- Complementarity Determining Region 3 Repertoire of Clonal T Cell Receptor α Chain in Patients with Micrometastasis from Breast Cancer
- 肿瘤防治研究, 2015, 42(11): 1109-1113
- Cancer Research on Prevention and Treatment, 2015, 42(11): 1109-1113
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2015.11.011
-
文章历史
- 收稿日期: 2014-10-28
- 修回日期: 2015-01-08
2. 450003 郑州,郑州大学附属肿 瘤医院 河南省肿瘤医院中心实验室;
3. 510089 广州,中 山大学中山医学院微生物学教研室;
4. 450003 郑州,郑州 大学附属肿瘤医院 河南省肿瘤医院血液科
2. Central Laboratory, The Affiliated Cancer Hospital of Zhengzhou University, He’nan Cancer Hospital, Zhengzhou 450003, China;
3. Department of Microbiology, Zhongshan Medical College, Sun Yat-sen University, Guangzhou 510089, China;
4. Department of Hematology, The Affiliated Cancer Hospital of Zhengzhou University, He’nan Cancer Hospital, Zhengzhou 450003, China
T细胞通过T细胞受体(T cell receptor, TCR)识别抗原,引起具有特定互补决定区3(complementarity determining region 3,CDR3)T细胞的克隆性增生,在机体抗肿瘤、抗感染和自身免疫性疾病中起着重要作用[1]。研究表明多种实体性肿瘤患者体内存在针对肿瘤相关抗原的T细胞克隆,分析这些克隆性T细胞及其分子特点,对于研究T细胞在肿瘤发生和发展中的变化,了解机体抗肿瘤T细胞免疫功能状态,发现疾病相关的T细胞克隆,开展个体化免疫治疗等具有重要意义[2]。本研究旨在利用改进的免疫扫描谱型技术分析乳腺癌微转移淋巴结中T细胞的克隆性增生,对单克隆增生T细胞扩增其全长编码序列,了解TCRα链可变区基因(TCR α chain variable region, TCR AV)、连接区基因(joining region, TCR AJ)所属家族选择性取用及CDR3序列特点,为寻找乳腺癌相关抗原表位等进一步的研究打下基础。
1 材料与方法 1.1 标本选取郑州大学附属肿瘤医院确诊为乳腺浸润性导管癌(组织学Ⅱ级)住院患者10例,均为未接受新辅助化疗的新发女性患者,年龄28~73岁。经术中快速冰冻检查明确腋窝前哨淋巴结有癌微转移新鲜淋巴结标本,淋巴结内癌巢最大直径≤2 mm且>0.2 mm者为微转移[3]。同时取5例反应性增生淋巴结新鲜标本作为对照。
1.2 主要试剂TRIzol试剂、SuperScript Ⅲ反转录酶、LD PCR引物购自美国Invitrogen公司;TaqDNA聚合酶、限制性核酸内切酶(XbaⅠ、EcoRⅠ、KpnⅠ)、T4 DNA连接酶、pGEM-7Zf(+)载体购自美国Promega公司;质粒小量提取试剂盒购自美国Omega公司;DNA胶纯化回收试剂盒购自德国QIAGEN公司。
1.3 RNA提取及cDNA合成送检淋巴结沿最大纵切面切开,冰冻切片诊断为癌微转移后对应HE切片中癌的位置,剪取淋巴结癌旁淋巴组织新鲜标本和对照新鲜淋巴结标本在200目筛网上研磨,用PBS进行冲洗,收集细胞悬液并计数,提取总RNA合成20 μl cDNA,LD PCR合成cDNA第二链(dsDNA)。
1.4 PCR扩增32个TCR AV亚家族根据文献[4]合成TCR AV32个家族上游引物34条,及共用FAM标记下游引物,引物由上海英俊生物技术有限公司合成。总反应体系50 μl,其中dsDNA模板1 μl,dNTP 5 μl,10×buffer 5 μl,25 mmol/L MgCl2 3 μl,上游引物1.8 μl,共用FAM标记的下游引物1.8 μl,Taq DNA聚合酶1.25 U,实验中不足体积用ddHl2O水补足。反应条件:95℃变性3 min;95℃ 30 s,60℃ 30 s,72℃ 90 s,35个循环;72℃延伸10 min。取PCR产物8 μl在1.5%琼脂糖凝胶中电泳。
1.5 基因扫描(GeneScan)分析取2 μl荧光引物扩增的PCR产物与3 μl DNA变性胶上样缓冲液混合,加入0.5 μl标准品,2 μl去离子甲酰胺,0.5 μl变性胶上样缓冲液,94℃变性4 min后上样至6%聚丙烯酰胺凝胶,373 DNA序列分析仪中电泳2 h,用GeneScan672软件进行基因扫描分析。
1.6 TCRα链全长编码序列扩增根据文献[5]合成扩增TCRα链各家族全长序列的上、下游引物,上游引物分别引入KpnⅠ和EcoRⅠ限制性内切酶识别序列,下游引物引入XbaⅠ限制性内切酶识别序列,将终止密码TGA改为TAA以避免甲基化。以单克隆增生家族的cDNA为模板,进行PCR扩增。总反应体系50 μl,其中10×Buffer 5 μl,MgCl2 3 μl,dNTP 1 μl,上、下游引物各0.5 μl,dsDNA 1 μl,Taq酶(5 u/μl)0.5 μl,实验中不足体积用ddHl2O水补足。各反应体系引物浓度是10 μmol/L,Mg2+终浓度1.5 mmol/L。反应条件:预变性95℃ 5 min;95℃ 40 s,< /span>60℃ 1 min,72℃ 1 min 50 s,35个循环;72℃延伸10 min。
1.7 TCRα链重组质粒构建及CDR3序列分析20 μl酶切反应体系:其中TCRα链PCR扩增产物10 μl,限制性内切酶XbaⅠ0.5 μl,EcoRⅠ或KpnⅠ0.5 μl,10×Buffer H 2 μl,乙酰BSA 0.2 μl,实验中不足体积用ddHl2O水补足。反应条件:37℃ 3 h。酶切后在1%琼脂糖凝胶中电泳并纯化回收,与相应酶切纯化回收的质粒pGEM-7Zf(+)在T4 DNA连接酶作用下16℃连接过夜,连接产物转化感受态细胞E. coli JM109,涂布于含氨苄青霉素的LB选择性平板进行筛选,挑取氨苄青霉素抗性菌落,置于含氨苄青霉素的LB液体培养液中,37℃振摇培养过夜,提取质粒并双酶切鉴定。阳性质粒委托上海英俊生物公司对TCRα链全长序列进行测序,利用DNAstar软件及IMGT TCR资源分析TCRα链CDR3序列及TCR AV、AJ所属亚家族。
2 结果 2.1 反应性增生、乳腺癌微转移淋巴结各家族RT-PCR结果乳腺癌微转移淋巴结与反应性增生淋巴结进行PCR扩增,结果显示对照组淋巴结全部32个亚家族均有扩增,扩增片段大小在150~500 bp之间,10例患者中7例的各家族均有扩增,病例2、3、7除1~3个家族无扩增外,其余家族均有扩增,病例1的32个AV家族RT-PCR扩增结果见图 1。

|
| AV: variable region of TCR α chain 图 1 病例1的32个TCR AV亚家族RT-PCR产物 Figure 1 RT-PCR products of TCR α chain of 32 AV subfamilies of case 1 |
2.2 反应性增生、乳腺癌微转移淋巴结TCR CDR3亚家族GeneScan分析结果
5例反应性增生淋巴结各家族基因扫描均呈现中间高两边低、多个峰的谱型,提示为多克隆。10例乳腺癌患者32个家族经基因扫描存在单峰、一主峰连同少数小峰、偏峰及多峰图像,提示PCR产物为单克隆、寡克隆、寡克隆趋势、偏移及多克隆。10个病例中有9个病例出现了单克隆和寡克隆或寡克隆趋势,其余为多克隆。病例9只出现偏峰及多克隆,无单克隆及寡克隆的优势表达。10个病例的基因扫描分析TCRAV亚家族表达分布及T细胞克隆特点,见表 1。病例1的TCR AV32个亚家族CDR3谱型GeneScan扫描分析图,见图 2。
 |

|
| 图 2 病例1 TCR AV32个亚家族CDR3谱型GeneScan扫描 分析图 Figure 2 Spectratyping of CDR3 sizes for 32 TCR AV subfamilies in case 1 |
将病例1、2、4、6、8共5例显示单克隆亚家族的TCRα链全长序列PCR扩增产物上1%琼脂糖凝胶电泳,扩增片段大小约800 bp,见图 3。阳性克隆酶切电泳可见两条泳动速度不同的条带,分别与TCRα链PCR扩增产物、空质粒酶切后的大小一致,见图 4。

|
| M: marker; 1-5: PCR results of case1, 2, 4, 6 and 8 图 3 病例1、2、4、6、8单克隆AV亚家族TCRα链全长序 列PCR产物 Figure 3 PCR products of TCR α chain complete DNA sequence (CDS) of monoclonal hyperplasia of AV subfamilies of case 1, 2, 4, 6 and 8 |
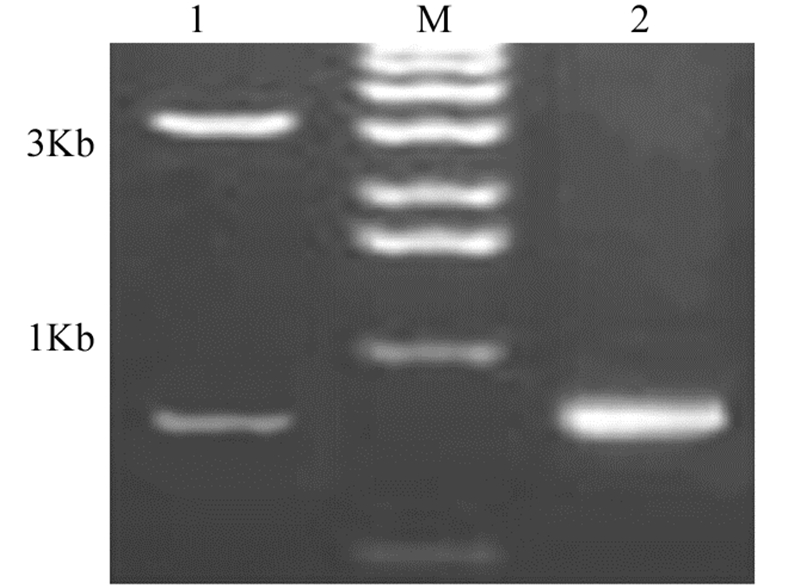
|
| 1: digestion with restriction endonucleases of recombinant plasmid of TCR α chain; 2: PCR result ; M: marker 图 4 TCRα链重组质粒PCR及酶切鉴定 Figure 4 Identification of recombinant plasmid of TCR α chain by PCR and digestion with restriction endonucleases |
2.4 TCRα链序列测定及CDR3谱型结果
5例单克隆增生AV家族经测序获得了TCRα链全长核苷酸序列。经分析比对克隆性T细胞取用的AV、AJ家族不完全相同,而且各克隆的氨基酸序列不同,长度在9~13个氨基酸之间,但是病例4和病例8具有相同的氨基酸基序:AM和DDKII,而且两个克隆的AV和AJ亚家族相同,均为AV12S3和AJ30。5个单克隆增生AV亚家族TCRα链AV、AJ亚家族及CDR3序列测定结果,见表 2。
 |
人类T细胞细胞膜上的TCR由α和β链或γ和δ链构成异源二聚体,根据异源二聚体的构成性质不同可将T细胞分为两群,即α/β和γ/δ T细胞,肿瘤相关的克隆性增殖T细胞主要为α/β T细胞。TCRα链由AV-AJ-AC基因片断重排后编码,构成具有不同特异性的TCR分子,AV-AJ基因片段进行重排和随机插入核甘酸(N区)形成一高度可变区,称为CDR3,由此决定T细胞识别抗原的多样性和特异性[1]。不同T细胞克隆CDR3长度和核苷酸序列不同,因此分析TCR CDR3长度和序列可作为判断T细胞克隆性的指标[6]。免疫扫描谱型技术[7]是一种基于上述理论,利用RT-PCR及GeneScan技术来确定T细胞克隆性,然后对出现单、寡克隆家族重新进行PCR扩增后测序,已广泛用于感染、肿瘤和自身免疫性疾病等T细胞克隆性及TCR分子特点的研究。
目前有关不同疾病T细胞克隆变化及TCR CDR3序列的分析主要针对TCRβ链,在感染性疾病、自身免疫性疾病中均发现T细胞的克隆性增生以及TCR BV亚家族的谱型偏移,但是在不同疾病T细胞克隆的研究中没有发现相同的CDR3氨基酸序列[8, 9]。随着TCRβ链CDR3谱系检测方法的成熟,研究者开始关注其和肿瘤的关系,在T细胞肿瘤及多种实体性肿瘤中也发现了T细胞的克隆性增生以及TCR BV亚家族的谱型偏移,不同克隆T细胞的出现可能与肿瘤细胞的异质性、肿瘤抗原或肿瘤相关抗原具有复杂性和多样性有关[10]。由于TCRα/β受MHC限制性作用下识别抗原时,需要α和β链的CDR3共同参与识别并与肿瘤相关抗原肽接触,因此对α/β TCR的研究应包括α和β链。在前期的研究中,我们分析了乳腺癌TCR BV亚家族的谱型偏移,不同患者仅表达3~5个Vβ亚家族,并且单/寡克隆增生的T细胞的CDR3序列不同[11]。根据研究需要,我们对传统免疫指纹技术进行了适当改进,合成TCRα链全长序列扩增引物,由原来扩增TCRV区部分序列改为扩增TCR全长编码序列,便于体外表达的需要,结合以往的研究为构建乳腺癌TCRα/β四聚体,用于建立乳腺癌新型分子检测试剂提供基础资料。
利用改进的免疫指纹技术,本研究对10例乳腺浸润性导管癌(组织学Ⅱ级)患者微转移淋巴结内T细胞的克隆性进行分析,结果与反应性增生淋巴结T淋巴细胞表达全部32个TCR AV亚家族不同,9例乳腺癌患者TCR AV亚家族表达存在明显的谱型偏移,出现一个或多个TCR AV亚家族的选择性取用,表明乳腺癌微转移淋巴结内存在针对乳腺癌细胞释放的肿瘤相关抗原诱导活化的T细胞克隆,并且优势增生的T细胞会影响其他家族T细胞的表达。病例9仅有峰型偏移,不存在T细胞的优势表达,这些峰型偏移也在另外9例患者中不同程度的出现。分析其原因可能是因为病变淋巴结中T细胞对肿瘤相关抗原的免疫反应处于一个动态的过程有关,也可能是因为机体免疫功能不健全,不能诱导出特异的T细胞免疫反应[12]。
对多克隆表达的TCRα链全长序列进行分析,5个序列具有部分共同的AV基因和相同的AC基因,其穿膜区基因也是相同的保守序列,各序列的CDR3区核苷酸和氨基酸序列却不相同,证实为不同的T细胞克隆,说明了乳腺癌相关抗原的多样性和复杂性。但是TCR识别抗原时除HLA型别差异影响外,和TCR CDR3的空间构型也存在密切关系,即不同谱型的CDR3如果空间结构相似很可能会识别相同的抗原表位[13],因此有必要结合TCRβ链研究克隆性T细胞TCRα/β空间结构特点。
本研究中病例4和病例8均优势表达TCR AV12S3和AJ30,而且病例8仅出现TCR AV12亚家族的单克隆增生,两个病例CDR3区含有相同的氨基酸基序:AM和DDKII克隆性T细胞TCRα链在基因重排时AV12S3和AJ30基因片段进行连接后插入了不同的碱基对,代表了不同的T细胞群。由于目前国内外对乳腺癌患者T细胞克隆性及TCR CDR3分子特征的研究报道较少[1],是否TCR AV12家族T细胞克隆是乳腺浸润性导管癌相关抗原的特异性效应T细胞,以及相同的氨基酸基序是否与乳腺癌特异性抗原具有一定相关性,还有待进一步研究,但是同一病例或不同病例之间发现相同或相似的CDR3氨基酸序列对于本研究具有重要意义,提示进一步扩大样本量或许对寻找乳腺癌相关抗原表位有所帮助,这也是目前许多研究者共同关心的问题。
总之,本研究通过分析乳腺癌淋巴结微转移患者标本中T细胞的克隆性增生及TCRα链谱型偏移,有助于了解机体抗肿瘤免疫功能状态,分析CDR3氨基酸序列,为研究乳腺癌肿瘤相关抗原表位,进行个体化免疫治疗等提供基础资料。
| [1] | He XY,Yang WM,Tang WT, et al. TRAV gene expression in PBMCs and TILs in patients with breast cancer analyzed by a DNA melting curve (FQ-PCR) technique for TCR α chain CDR3 spectratyping[J]. Neoplasma, 2012, 59(6): 693-9. |
| [2] | Sherwood AM, Emerson RO, Scherer D, et al. Tumor-infiltrating lymphocytes in colorectal tumors display a diversity of T cell receptor sequences that differ from the T cells in adjacent mucosal tissue[J]. Cancer Immunol Immunother, 2013, 62(9): 1453-61. |
| [3] | Grabau D, Ryden L, Fern? M, et al. Analysis of sentinel node biopsy-a single-institution experience supporting the use of serial sectioning and immunohistochemistry for detection of micrometastases by comparing four different histopathological laboratory protocols[J]. Histopathology, 2011, 59(1): 129-38. |
| [4] | Yao XS, Ma L, Wen Q, et al. Establish and identify the immune spectratyping analysis to monitor the TCR CDR3 Spectatypiny[J].Zhonghua Sheng Wu Xue He Mian Yi Xue Za Zhi, 2006, 26(6): 571-3. [姚新生, 马骊, 温茜, 等. 监测TCR CDR3漂移的免疫扫描谱型分析技术的建立与鉴定[J]. 中华微生物学和免疫学杂志, 2006, 26(6): 571-3.] |
| [5] | Zhang JB, Fang YM, Huang Y, et al. Analyses for the α/β T Cell Receptor Gene Rearrangement and CDR3 Repertoire in Active Pulmonary Tuberculosis Patients[J]. Zhongguo Mian Yi Xue Za Zhi, 2007, 23(12): 1136-9. [张建波, 方毅敏, 黄艳, 等. 活动性肺结核患者α/β TCR基因重排及CDR3谱型分析[J]. 中国免疫学杂志, 2007, 23(12): 1136-9.] |
| [6] | Fujii Y, Hayasaka D, Kitaura K, et al. T-cell clones expressing different T-cell receptors accumulate in the brains of dying and surviving mice after peripheral infection with far eastern strain of tick-borne encephalitis virus[J]. Viral Immunol, 2011, 24(4): 291-302. |
| [7] | Blohm JH, Blohm N, Hummel M, et al. Detection of clonal T-cell-receptor (TCR) Vbeta rearrangements in explanted dilated cardiomyopathy hearts by semi-nested PCR,GeneScan, and direct sequencing[J]. Med Sci Monit Basic Res, 2013, 19: 111-7. |
| [8] | Russi S, Lauletta G, Serviddio G. T cell receptor variable β gene repertoire in liver and peripheral blood lymphocytes of chronically hepatitis C virus-infected patients with and without mixed cryoglobulinaemia[J]. Clin Exp Immunol, 2013, 172(2): 254-62. |
| [9] | Zou HY, Yu WZ, Zhang Q, et al. Preliminary study of the clonal characteristics of the TCR BV subfamilies in T cells in the peripheral blood from patients with uveitis[J]. Genet Mol Res, 2014, 13(1): 1296-303. |
| [10] | Clemente MJ, Wlodarski MW, Makishima H, et al. Clonal drift demonstrates unexpected dynamics of the T-cell repertoire in T-large granular lymphocyte leukemia[J]. Blood, 2011, 118(16): 4384-93. |
| [11] | Zhang JB, Song YP, Yu QK, et al. Analysis of the TCR β-chain gene rearrangement and CDR3 repertoire in patients with breast cancer[J]. Zhongliu Yan Jiu Yu Lin Chuang, 2010, 22(3), 179-81. [张建波, 宋永平, 于庆凯, 等. 乳腺癌患者T细胞受体基因重排及CDR3区序列分析[J]. 肿瘤研究与临床, 2010, 22(3), 179-81.] |
| [12] | Luo W, Liao WJ, Huang YT, et al. Cancer of the gastrointestinal tract results in a restricted T-cell repertoire dependent upon tumor differentiation[J]. Cell Immunol, 2011, 270(1): 47-52. |
| [13] | Yin QS, Tan H, Chen SH, et al. Cloning of antigen-specific TCRα and β genes and their 3-D structure prediction [J]. Di San Jun Yi Da Xue Xue Bao, 2011, 33(4): 349-52. [尹青松, 谭获, 陈少华, 等. 抗原特异TCRα和β链基因克隆及分子空间结构预测[J]. 第三军医大学学报, 2011, 33(4): 349-52.] |
 2015, Vol. 42
2015, Vol. 42


