文章信息
- 赵连梅, 冯佳, 李燕, 贾楠, 戴素丽, 冯强, 单保恩.
- ZHAO Lianmei, FENG Jia, LI Yan, JIA Nan, DAI Suli, FENG Qiang, SHAN Baoen.
- 胃癌研究中实时定量PCR实验内参基因的选择
- Optimization of Housekeeping Gene for Normalization of Quantitative Polymerase Chain Reaction Assay in Gastric Cancer Research
- 肿瘤防治研究, 2015, 42(10): 974-978
- Cancer Research on Prevention and Treatment, 2015, 42(10): 974-978
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2015.10.005
-
文章历史
- 收稿日期: 2014-09-28
- 修回日期: 2014-12-30
2. 050032石家庄,白求恩国际和平医院胃肠科;
3. 100021北京,中国医学科学院肿瘤医院胸外科
2. Department of Gastroenterology, Bethune International Peace Hospital of Chinese PLA, Shijiazhuang 050082, China;;
3. Department of Abdominal Surgical Oncology, Cancer Hospital, Chinese Academy of Medical Science, Beijing 100021, China
反转录实时定量聚合酶链技术(real time quantitativereverse transcription-polymerase chain reaction,qRT-PCR)具有定量准确、敏感度高和高通量等特点[1, 2],已被广泛应用于基因表达的检测分析中,尤其是近年来基因组中非编码RNA(noncodingRNA)的发现,其数量远远大于编码RNA,但由于其不能有效编码蛋白而不能进行蛋白的表达检测,从而使qRT-PCR技术成为检测基因表达量最为简便易行的手段之一[3]。在进行实时定量PCR实验时需要应用内参基因对目标基因的表达量进行校正,才能获得真实而可靠的结果。而这些内参基因往往由看家基因(housekeeping gene)充当,看家基因有多种,最为常用的为GAPDH、ACTB、18sRNA、28sRNA、RPⅡ和Tublin等基因[4, 5]。理想的内参基因应该是在各种实验因素影响和在各种类型的组织或细胞中均恒定表达[6]。然而,很多实验结果表明,任何内参基因的恒定表达都只是在一定类型的组织和细胞中,或一定的实验因素作用下的恒定,在其他类型的组织细胞或实验因素作用下则是变化的[7, 8, 9, 10]。胃癌是世界上排名第四位的癌症,在癌症死亡率中排名第二位[11]。明确胃癌发生发展的机制,发现其早期诊断的分子标志物和治疗靶点尤为重要。筛选胃癌肿瘤标志物最常用的手段有高通量芯片技术和qRT-PCR技术,这些技术都要求有稳定的看家基因作为内参基因。本研究旨在寻找在胃癌和癌旁组织、胃上皮永生化细胞和胃癌细胞中表达比较稳定的内参基因,为差异表达基因的筛选提供实验基础。
1 材料与方法 1.1 材料本研究所用50例胃癌组织(术后均经病理诊断为胃腺癌)和癌旁组织标本(距癌组织边缘≥5 cm)均取自河北医科大学第四医院2012年4月—12月住院的胃癌患者手术切除标本,且术前未进行放、化疗或其他治疗。人正常胃黏膜上皮细胞系GSE-1由北京肿瘤防治研究中心遗传室柯杨教授[12]惠赠。胃癌细胞系MGC-803、SGC-7901、MKN-28、HGC-27由中国医学科学院基础医学研究所细胞中心提供。
1.2 试剂和仪器总RNA提取试剂TRIzol购自美国Invitrogen公司,反转录试剂盒购自美国Promega公司。PCR引物由北京中美泰合生物公司负责合成和纯化,用无菌去离子水稀释,引物终浓度为10 μmol/L,序列见表 1。荧光定量PCR酶使用Takara公司的2×SYBgreen mix。荧光定量PCR仪为ABI公司的9700HT Stepone plus,分光光度计为NanoDrop ND-2000c(Thermo,美国)。
TRIzol法提取组织总RNA,采用NanoDrop ND-2000c分光光度计测定RNA浓度,1%琼脂糖凝胶电泳检测RNA完整性。按反转录试剂盒说明书进行总RNA的反转录。反转录条件为:70℃ 5 min;4℃ 5 min;42℃60 min;72℃ 15 min;4℃停止转录。
1.3.2 qRT-PCR扩增qRT-PCR反应总体系为10 μl,其中2×SYBgreen mix 5 μl,cDNA 1 μl,去离子水3.6 μl,上下游引物(10 pmol/μl)各0.2 μl。引物参看文献或者用primer5设计,序列见表 1。反应条件:95℃ 5 min;95℃ 30 s,60℃ 30 s,72℃ 30 s(40个循环),70℃到99℃每度8个荧光采集点绘制溶解曲线。通过观察融解曲线是否为单一峰评价PCR反应产物的特异性。每个反应设三个复孔,复孔间Ct值小于0.5认为有意义。
1.3.3 geNorm程序统计学分析下载geNorm程序,在Excel表中设定不同样本中某一看家基因Ct值最小者的表达量为1,其他样本此看家基因的相对表达量为2-ΔΔCt(ΔCt=各样本Ct值-最小Ct值),将这些数据导入geNorm程序。利用该程序计算基因表达稳定度的平均值M,对看家基因的表达稳定度进行排序(M值越小,表达越稳定)。
1.4 统计学方法所有数据均采用SPSS17.0统计软件进行分析,首先对实验数据进行正态分布检测,如符合正态分布,使用双尾配对t检验,不符合正态分布使用秩和检验。所有数值均用均数+标准差表示,P<0.05为差异有统计学意义。
2 结果 2.1 GAPDH、ACTB、RPⅡ和18sRNA在胃癌组织中的表达分别提取50例胃癌和对应癌旁组织总RNA,进行精确定量后行qRT-PCR实验。结果发现,与癌旁对照组相比,GAPDH在胃癌组织中上调2.70±0.41个循环和(6.49±1.32)倍 ,ACTB上调2.33±0.32个循环和(5.0±1.24)倍,RPⅡ上调(1.88±0.21)个循环和(3.68±1.15)倍,差异均有统计学意义(P<0.05)。18sRNA循环数差异为(0.02±0.00)个循环,两组比较差异无统计学意义(P>0.05),见图 1。

|
| A: total cycle of reference genes expression in cancer tissue and non-cancer tissues detected by qRT-PCR, Ct=mean+SD; *: P<0.05, compared between two groups; B: melting curves of GAPDH, ACTB, RPⅡ and 18sRNA in real-time PCR test; C: Expression change of housekeeping genes in cancer tissue and corresponding adjacent tissues 图 1 qRT-PCR检测内参基因GAPDH、ACTB、RPII和18sRNA的实时表达量 Figure 1 Real-time expression of GAPDH, ACTB, RPII and 18sRNA determined by qRT-PCR |
正常胃黏膜上皮细胞GSE-1及四种胃癌肿瘤细胞系(MGC-803、SGC-7901、MKN-28和HGC-27)的qRT-PCR实验结果显示,GAPDH在五种细胞系中的Ct值分别为(16.72±1.13)、(16.10±0.85)、(15.8±1.32)、(15.9±1.10)和(17.2±2.10),ACTB在五种细胞系中的Ct值分别为(15.2±0.54)、(15.4±1.41)、(15.4±1.62)、(15.5±1.70)和(15.7±0.73),RPⅡ在五种细胞系中的Ct值分别为(22.3±2.01)、(23.1±1.84)、(23.7±2.03)、(22.6±2.65)、(22.4±2.21)。18sRNA在五种细胞系中的Ct值分别为(14.7±2.61)、(14.1±1.78)、(13.1±0.57)、(12.6±1.31)、(11.5±1.20),各组中肿瘤细胞Ct值与胃正常上皮细胞GSE-1的Ct值相比,差异均无统计学意义(P>0.05),见图 2,我们还发现RPⅡ在五种细胞系中的Ct值均高于22个循环,相较于其他内参基因,表达较低。
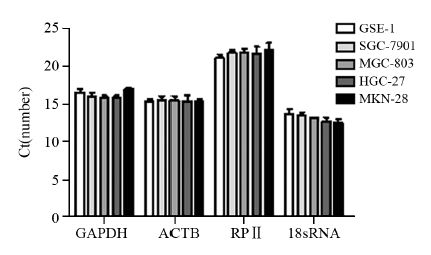
|
| 图 2 qRT-PCR实验检测GAPDH、ACTB、RPⅡ和 18sRNA内参基因在正常胃上皮细胞和胃癌细胞中的表达 Figure 2 Expression of GAPDH, ACTB, RPⅡ and 18sRNA determined by qRT-PCR in normal gastric epithelial cells and gastric cancer cells |
以GAPDH、ACTB、RPⅡ和18sRNA分别作内参时,检测CEA在50例胃癌和癌旁组织中的表达。结果显示,以GAPDH作为内参时,CEA在癌组织中的表达呈下降趋势,下降的倍数为(1.71±0.38)倍,以ACTB和RPⅡ作为内参时,CEA在癌和癌旁组织中未发生明显的变化,以18sRNA作为内参时,CEA在胃癌组织中明显上升(P<0.05),上升的倍数为(2.67±0.51)倍,见图 3。

|
| *: P<0.05, compared between two groups 图 3 qRT-PCR检测以GAPDH、ACTB、RPII和18sRNA 分别作为内参基因qRT-PCR时CEA基因的相对表达量 Figure 3 Comparative Expression of CEA as Determined by qRT-PCR for Four Housekeeping Genes GAPDH, ACTB, RPⅡ and 18sRNA |
为了验证上述实验结果,使用geNorm程序及用50例胃癌和癌旁组织中内参基因的表达量来选择最适内参。在本研究体系中,GAPDH、ACTB、RPⅡ和18sRNA表达稳定度的平均值M依次为0.274,0.235,0.168,0.106,依照M值越小越稳定的原则,18sRNA在胃癌和癌旁体系中表达稳定性最高,而GAPDH的稳定性最低,见图 4。

|
| 图 4 四种内参基因的平均表达稳定性 Figure 4 Average expression stability of four housekeeping genes |
随着高通量基因研究技术日新月异的发展,研究者在基因组中发现了大量除mRNA以外的非编码RNA,近年研究证实这些非编码RNA对生命体的发生和发展及疾病的发生发挥着重要的作用。在非编码RNA研究中,第一步就是利用合适的实验手段鉴定差异基因的表达。因非编码RNA不具备编码蛋白的功能,因此对其检测手段较为局限,实时定量PCR(real time quantitative reversetranscription-polymerase chain reaction,qRT-PCR)就成为检测其表达水平最简便易行的方法之一。qRT-PCR实验技术能够以高敏感度和高通量的优点快速鉴别出组间基因表达的差异。但是首先需要应用内参基因对目标基因表达量进行校正,而选择合适的看家基因作为内参照基因对于差异RNA的筛选至关重要[12, 13]。所谓内参照基因即要求此基因在所有的细胞和组织中恒定表达,而不受任何处理因素的影响。但事实上绝对不发生变化的基因是不存在的,因此在实验时,要选择本体系中变化最小、表达最为稳定的基因作为内参。也有很多研究者提出了可以用总RNA来进行样品组间的标准化,但是这种方法不能避免由反转录及PCR反应等步骤引起的实验误差,另外,非常准确而可靠的RNA定量方法也是很难找到的。核酸定量仪和分光光度计法定量检测RNA都存在敏感度较低、易受残存DNA的影响,较低浓度的样品定量也不准确等,并且总RNA作内参不能校正反转录效率差异等引起的实验误差。因此合适的、表达较为恒定的看家基因作为内参基因是较为理想的手段。
本研究选择了胃癌研究中常用的内参基因GAPDH、ACTB、RPⅡ和18sRNA作为研究对象分析其在组织和细胞中的表达。研究发现,四种看家基因中GAPDH、ACTB在胃癌和癌组织中中度表达,RPⅡ表达水平最低,18sRNA表达最高。在50例标本中,胃癌组织与相对应的癌旁组织相比,GAPDH、ACTB的表达水平明显增高,其中GAPDH在78%的组织标本中表达升高,ACTB在74%的组织标本中表达升高,RPⅡ在52%的组织标本中表达升高。而18sRNA在胃癌组织与癌旁组织中未发生明显的表达差异。根据内参基因需要相对恒定表达的原则,18sRNA最适宜作为胃癌研究中实时定量PCR实验的内参基因。那么作为内参基因的GAPDH、ACTB、RPⅡ为何在癌组织和癌旁组织中具有表达差异呢?我们推测组织的不均一性是其重要原因,肿瘤组织中存在各种间质细胞,包括成纤维细胞、淋巴细胞等,此类细胞为非上皮细胞,可能内参基因的表达水平在上皮细胞和间质细胞中存在差异,但仍需要进行验证。本研究还发现四种内参基因在癌细胞和正常细胞中恒定表达,推测正常胃黏膜上皮及胃癌细胞均为腺上皮细胞,内参基因的表达比较恒定。但是RPⅡ在胃癌细胞和胃永生化上皮细胞中表达量较低,不适宜作为内参基因。因此GAPDH、ACTB和18sRNA可作为胃癌细胞研究中的内参基因。
为了验证18sRNA在胃癌研究中作为内参基因的可行性,我们将四种内参基因作为内参基因分别进行胃癌组织中CEA表达的检测,结果发现以GAPDH、ACTB和RPⅡ作为内参对照基因时,胃癌肿瘤标志物基因CEA在胃癌组织中并未发生明显的变化,而以18sRNA作为内参对照基因时,CEA在胃癌组织中的表达明显升高,说明内参基因选用不合适,将会导致实验结果与事实不相符。另外,样本的数量是组织中基因表达检测的另一项重要指标,由于个体差异和肿瘤本身异质性等原因,组织样本需要一定数量才可以尽量减少实验误差。总之,内参基因的选择对于目标基因表达的准确定量(尤其是新物种基因表达研究)至关重要。Vandesompele和Katleen等于2002年编写的geNorm程序广泛用于内参基因的选择。研究者可使用geNorm程序,从一系列不同功能的候选看家基因中选择出最适宜的看家基因作为内参基因用于目标基因的标准化,从而达到准确定量和筛选的实验目的。
| [1] | Huggett J, Dheda K, Bustin S, et al. Real-time RT-PCR ormalisation; strategies and considerations[J]. Genes Immun, 005, 6(4): 279-84. |
| [2] | Radoni? A, Thulke S, Mackay IM, et al. Guideline to reference ene selection for quantitative real-time PCR[J]. Biochem iophys Res Commun, 2004, 313(4): 856-62. |
| [3] | Mattick JS. The genetic signatures of noncoding RNAs[J]. PLoS enet, 2009, 5(4): e1000459. |
| [4] | Vandesompele J, De Preter K, Pattyn F, et al. Accurate ormalization of real-time quantitative RT-PCR data by geometric veraging of multiple internal control genes[J]. Genome Biol, 002, 3(7): RESEARCH0034 34. |
| [5] | Koon N, Schneider-Stock R, Sarlomo-Rikala M, et al. Molecular argets for tumour progression in gastrointestinal stromal umours[J]. Gut, 2004, 53(2): 235-40. |
| [6] | Liu S, Zhu P, Zhang L, et al. Selection of reference genes for T-qPCR analysis in tumor tissues from male hepatocellular arcinoma patients with hepatitis B infection and cirrhosis[J]. ancer Biomark, 2013, 13(5): 345-9. |
| [7] | Goidin D, Mamessier A, Staquet MJ, et al. Ribosomal 18S RNA revails over glyceraldehyde-3- phosphate dehydrogenase and eta-actin genes as internal standard for quantitative comparison f mRNA levels in invasive and noninvasive human melanoma ell subpopulations[J]. Anal Biochem, 2001, 295(1): 17-21. |
| [8] | Hamalainen HK, Tubman JC, Vikman S, et al. Identification and alidation of endogenous reference genes for expression profiling f T helper cell differentiation by quantitative real-time RTPCR[ ]. Anal Biochem, 2001, 299(1): 63-70. |
| [9] | Glare EM, Divjak M, Bailey MJ, et al. beta-Actin and GAPDH ousekeeping gene expression in asthmatic airways is variable nd not suitable for normalizing mRNA levels[J]. Thorax, 2002, 7(9): 765-70. |
| [10] | Steele BK, Meyers C, Ozbun MA. Variable expression of ome ‘‘housekeeping’’ genes during human keratinocyte ifferentiation[J]. Anal Biochem, 2002, 307(2): 341-7. |
| [11] | Jemal A, Siegel R, Xu J, et al. Cancer statistics, 2010[J]. CA ancer J Clin, 60(5): 277-300. |
| [12] | Wang BJ, Zhang ZQ, Ke Y. Conversion of cadherin isoforms in ultured human gastric carcinoma cells[J]. World J Gastroenterol, 006, 12(6): 966-70. |
| [13] | Zhang Q, Geng PL, Yin P, et al. Down-regulation of long noncoding NA TUG1 inhibits osteosarcoma cell proliferation and romotes apoptosis[J]. Asian Pac J Cancer Prev, 2013, 14(4): 311-5. |
 2015, Vol. 42
2015, Vol. 42



