文章信息
- 徐若冰,薛恒川,王建平,吴亮,王建明. 2015.
- XU Ruobing, XUE Hengchuan, WANG Jianping, WU Liang, WANG Jianming. 2015.
- 食管鳞状细胞癌差异DNA甲基化位点初筛及异常甲基化谱的构建
- Screening of Differential DNA Methylation Profile and Construction of Aberrant Methylation Panel in Esophageal Squamous Cell Carcinoma
- 肿瘤防治研究, 2015, 42(01): 28-31
- Cancer Research on Prevention and Treatment, 2015, 42(01): 28-31
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2015.01.007
-
文章历史
- 收稿日期:2013-12-02
- 修回日期:2014-01-22
2. 212200 扬中,江苏省扬中市人民医院胸外科;
3. 211166 南京,南京医科大学公共卫生学院社会医学与健康教育学系
2. Department of Thoracic Surgery, People’s Hospital of Yangzhong, Yangzhong 212200, China;
3. Department of Social Medicine and Health
表观遗传学变化是指在核苷酸序列不变的情 况下,基因表达发生可遗传的改变,如DNA甲基 化、组蛋白乙酰化、RNA干扰等。在许多真核生 物中,这是一种基因组用于抵制外源性寄生基因 片段侵袭的保护机制[1]。作为表观遗传改变的主要 形式,DNA甲基化是哺乳动物遗传外修饰的重要 调控方式,启动子或外显子区CG岛异常甲基化可 导致基因表达沉默、功能丧失和肿瘤发生。
基因组DNA异常甲基化是肿瘤发生发展过程 中的一个早期事件,因而在早期诊断和预后方面 具有广阔的应用前景[2, 3]。近年来DNA甲基化在医 学研究领域倍受关注,在研究方法上也取得突破 性进展,基因芯片的研发为大规模检测海量位点 提供了可能。以Illumina公司的450K甲基化芯片为 例,该芯片设计了485 577个甲基化位点,覆盖了 96%的CG岛,常被用于前期特异性基因和位点的 筛选。为探讨其在构建食管鳞状细胞癌异常甲基 化谱中的作用,我们进行了该项研究。 1 资料与方法 1.1 芯片初筛
从上消化道癌高发区江苏省扬中市选择5例 经病理学确诊的食管鳞状细胞癌患者。手术时分 别留取癌灶中心和切口远端肉眼正常食管黏膜组 织,低温冻存。同时在参加当地社区早期癌症筛 查的人群中留取2例正常食管黏膜组织为对照。 组织经研磨、匀浆后提取基因组DNA,亚硫酸氢 盐转化后经过扩增、断裂、沉淀、重悬,在450K 甲基化芯片上经过杂交、洗涤、延伸、染色、扫 描等过程,获得DNA甲基化信号。信号采集与分 析采用GenomeStudio®甲基化分析系统。针对每个 CG位点设计两种探针,用双色荧光信号检测甲基 化和非甲基化等位基因,采用杂交质控图和背景 图来检测实验效果。差异甲基化基因的筛选标准 为:实验组与对照组其中任何一组中为有效基因 (P<0.05),且实验组样本差异分值(Diffscore) 小于-13或大于13,甲基化β差值(Delta Beta)大于 0.17或小于-0.17。差异分值大于13且甲基化β差值 大于0.17为差异甲基化位点。 1.2 质谱法验证
另取11例食管鳞状细胞癌灶中心组织和3例正 常对照,采用Sequenom质谱法对初筛出的位点进 行验证。基本步骤包括:亚硫酸盐处理;PCR扩 增甲基化区域片段;虾碱性磷酸酶反应;碱基特 异性酶切反应;纯化;点样;质谱芯片检测;数 据文件导出与分析。 2 结果 2.1 差异甲基化位点与基因组注释区域的分布
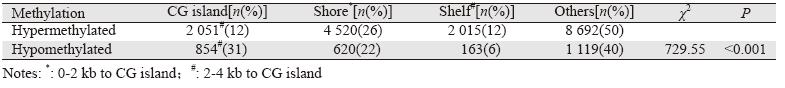
芯片检测的485 577个位点对应365 860个基因, 根据甲基化β差值和差异分值的筛选条件,食管鳞状 细胞癌组织和正常食管黏膜组织存在约4%差异甲基 化位点,高甲基化位点主要分布在基因体和启动子 区,低甲基化位点主要分布在启动子区和其他区域, 见表 1。平均每个高甲基化位点对应1.6个功能性位 点;1个低甲基化位点对应2.2个功能性位点。启动子 区的高甲基化位点主要分布在转录起始位点1 500 bp 和5’非翻译区(UTR),低甲基化位点主要在转录起 始位点200 bp和5’UTR,见表 2。CG岛周围区域也有 高甲基化现象,根据CG含量和距离远近分为岛滩区 和岛架区。高甲基化位点主要分布在CG岛滩区,低甲 基化位点主要分布在CG岛,见表 3。
 |
 |
 |
根据食管鳞状细胞癌组织和正常食管黏膜对照 间甲基化差异值,结合Genecards、Intogen等数据库筛 选出用于后续验证的候选异常高甲基化基因,见表 4。
 |
选择AIM2和CASZ1基因采用质谱法验证,结 果显示AIM2基因的3个甲基化位点甲基化频率在病 例和对照中的差异均无统计学意义。芯片检测中 发现的CASZ1_CpG_5位点及其周围的多个CG位点 的甲基化频率均表现为肿瘤组织高于正常对照, 见表 5。
 |
食管鳞状细胞癌早期多无明显症状与体征, 一旦确诊大都已是中晚期,患者预后不佳。因此 寻找敏感、特异的早期生物标志物对于食管鳞状 细胞癌早期诊断具有重要价值。甲基化芯片有助 于从海量的CG位点中筛选出有潜在应用价值的基因,然后进行评价并最终实现临床转化。
本研究发现,食管鳞状细胞癌组织异常高甲 基化位点主要分布在基因体,低甲基化位点主要 分布在启动子区。启动子区高甲基化位点主要在 TSS1500和5’UTR区域,低甲基化位点主要在启动 子的TSS200和5’UTR。近年研究发现,在CG岛和 岛滩外的其他区域也有分布,且这些区域的甲基 化与基因表达也存在关联[4]。本研究结果显示,发 生在岛滩的高甲基化位点多于CG岛。既往研究发 现,岛滩区甲基化程度在不同癌症患者中不一致[5], 与基因表达的关系可能比CG岛更强[6]。
通过芯片技术初筛出10个具有潜在应用价值的 差异甲基化基因,这些基因涉及众多的肿瘤相关生 物学过程。例如,ING2是生长抑制剂家族一员, 与细胞生长停滞、凋亡和DNA修复有关,可以激 活p53及其介导的凋亡通路,是一个重要的抑癌基 因[7],其表达缺失和头颈部鳞癌[8]和黑色素瘤[9]有 关。AIM2和炎性反应相关,能够抑制核因子和增 加G1期细胞群,诱导细胞凋亡,抑制乳腺肿瘤细胞 的生长[10]。CDH13是钙黏蛋白家族成员,在食管 鳞状细胞癌组织中的表达量明显低于癌旁和正常 食管黏膜组织[11]。KLF6是一个锌指肿瘤抑制子, 涉及到细胞分化和发育以及生长信号转导、细胞 凋亡和血管生成,既往研究发现与胃癌[12]、前列腺 癌[13]、以及大肠癌[14]相关。TRIT1负责tRNA的异戊 酰基转移,该基因与肺癌的发生有关[15]。TMEFF2 高甲基化在胶质瘤、卵巢癌、直肠癌、结肠癌和 肺癌中均有报道[16],且在食管鳞状细胞癌组织中 甲基化程度显著高于癌旁组织,去甲基化后mRNA 的表达增加[17]。CASZ1是与神经母细胞瘤有关的 候选抑癌基因[18]。ADAMTS9是金属蛋白酶家族成 员,与鼻咽癌及淋巴结转移有关[19],在胃癌、大肠 癌、胰腺癌中均存在高甲基化现象[20],研究表明发 现ADAMTS9的抑癌功能和抑制血管生成有关[21]。 EBF3能诱导细胞周期停滞和凋亡[22],可能参与转 化生长因子β信号转导通路。IQGAP2是含IQ基团 的GTP酶激活蛋白2,在细胞黏附、迁移、侵袭和 癌细胞转移中起着举足轻重的作用,在原发性胃癌 中,有47%的患者被检测到异常高甲基化[23],在肝癌[24]和前列腺癌[25]中可观察到其表达缺失。
基因芯片在技术可靠性得到保证的基础上, 具有高敏感度、高通量的特点,但由于价格因素 和数据处理、挖掘困难,目前尚未广泛应用于常 规样本检测,多用于前期筛选。基于芯片初筛获 得的差异甲基化位点,需要进一步扩大样本进行 验证和评估。本研究中,芯片初筛发现的两个差 异甲基化位点有1个在质谱法验证中仍具有统计学 意义。提示构建应用于食管鳞状细胞癌诊断与预 后的异常甲基化谱需经过不同人群、大样本、多 阶段的验证和评价,才能最终实现向临床应用的 转化。
| [1] | Laird PW. Cancer epigenetics[J]. Hum Mol Genet, 2005, 14 SpecNo 1: R65-76. |
| [2] | Kim YH, Lee HC, Kim SY, et al. Epigenomic analysis ofaberrantly methylated genes in colorectal cancer identifies genescommonly affected by epigenetic alterations[J]. Ann Surg Oncol,2011, 18(8): 2338-47. |
| [3] | Shah AK, Saunders NA, Barbour AP, et al. Early diagnosticbiomarkers for esophageal adenocarcinoma--the current stateof play[J]. Cancer Epidemiol Biomarkers Prev, 2013, 22(7):1185-209. |
| [4] | Jjingo D, Conley AB, Yi SV, et al. On the presence and role ofhuman gene-body DNA methylation[J]. Oncotarget, 2012, 3(4):462-74. |
| [5] | Doi A, Park IH, Wen B, et al. Differential methylation of tissueandcancer-specific CpG island shores distinguishes humaninduced pluripotent stem cells, embryonic stem cells andfibroblasts[J]. Nat Genet, 2009, 41(12): 1350-3. |
| [6] | Ji H, Ehrlich LI, Seita J, et al. Comprehensive methylome map oflineage commitment from haematopoietic progenitors[J]. Nature,2010, 467(7313): 338-42. |
| [7] | Okano T, Gemma A, Hosoya Y, et al. Alterations in novelcandidate tumor suppressor genes, ING1 and ING2 in human lungcancer[J]. Oncol Rep, 2006, 15(3): 545-9. |
| [8] | Borkosky SS, Gunduz M, Nagatsuka H, et al. Frequent deletionof ING2 locus at 4q35.1 associates with advanced tumor stagein head and neck squamous cell carcinoma[J]. J Cancer Res ClinOncol, 2009, 135(5): 703-13. |
| [9] | Lu F, Dai DL, Martinka M, et al. Nuclear ING2 expression isreduced in human cutaneous melanomas[J]. Br J Cancer, 2006,95(1): 80-6. |
| [10] | Chen IF, Ou-Yang F, Hung JY, et al. AIM2 suppresses humanbreast cancer cell proliferation in vitro and mammary tumorgrowth in a mouse model[J]. Mol Cancer Ther, 2006, 5(1): 1-7. |
| [11] | Jin Z, Cheng Y, Olaru A, et al. Promoter hypermethylationof CDH13 is a common, early event in human esophagealadenocarcinogenesis and correlates with clinical risk factors[J].Int J Cancer, 2008, 123(10): 2331-6. |
| [12] | Cho YG, Kim CJ, Park CH, et al. Genetic alterations of the KLF6gene in gastric cancer[J]. Oncogene, 2005, 24(28): 4588-90. |
| [13] | Narla G, Heath KE, Reeves HL, et al. KLF6, a candidate tumorsuppressor gene mutated in prostate cancer[J]. Science, 2001,294(5551): 2563-6. |
| [14] | Reeves HL, Narla G, Ogunbiyi O, et al. Kruppel-like factor 6(KLF6) is a tumor-suppressor gene frequently inactivated incolorectal cancer[J]. Gastroenterology, 2004, 126(4): 1090-103. |
| [15] | Spinola M, Galvan A, Pignatiello C, et al. Identification andfunctional characterization of the candidate tumor suppressor geneTRIT1 in human lung cancer[J]. Oncogene, 2005, 24(35): 5502-9. |
| [16] | Lin K, Taylor JR Jr, Wu TD, et al. TMEFF2 is a PDGF-AAbinding protein with methylation-associated gene silencing inmultiple cancer types including glioma[J]. PLoS One, 2011, 6(4):e18608. |
| [17] | Tsunoda S, Smith E, De Young NJ, et al. Methylation of CLDN6,FBN2, RBP1, RBP4, TFPI2, and TMEFF2 in esophagealsquamous cell carcinoma[J]. Oncol Rep, 2009, 21(4): 1067-73. |
| [18] | Liu Z, Yang X, Li Z, et al. CASZ1, a candidate tumor-suppressorgene, suppresses neuroblastoma tumor growth throughreprogramming gene expression[J]. Cell Death Differ, 2011,18(7): 1174-83. |
| [19] | Lung HL, Lo PH, Xie D, et al. Characterization of a novelepigenetically-silenced, growth-suppressive gene, ADAMTS9,and its association with lymph node metastases in nasopharyngealcarcinoma[J]. Int J Cancer, 2008, 123(2): 401-8. |
| [20] | Zhang C, Shao Y, Zhang W, et al. High-resolution meltinganalysis of ADAMTS9 methylation levels in gastric, colorectal,and pancreatic cancers[J]. Cancer Genet Cytogenet, 2010, 196(1):38-44. |
| [21] | Lo PH, Lung HL, Cheung AK, et al. Extracellular proteaseADAMTS9 suppresses esophageal and nasopharyngeal carcinomatumor formation by inhibiting angiogenesis[J]. Cancer Res, 2010,70(13): 5567-76. |
| [22] | Zhao LY, Niu Y, Santiago A, et al. An EBF3-mediatedtranscriptional program that induces cell cycle arrest andapoptosis[J]. Cancer Res, 2006, 66(19): 9445-52. |
| [23] | Jin SH, Akiyama Y, Fukamachi H, et al. IQGAP2 inactivationthrough aberrant promoter methylation and promotion of invasionin gastric cancer cells[J]. Int J Cancer, 2008, 122(5): 1040-6. |
| [24] | White CD, Khurana H, Gnatenko DV, et al. IQGAP1 andIQGAP2 are reciprocally altered in hepatocellular carcinoma[J].BMC Gastroenterol, 2010, 10: 125. |
| [25] | Xie Y, Yan J, Cutz JC, et al. IQGAP2, A candidate tumoursuppressor of prostate tumorigenesis[J]. Biochim Biophys Acta,2012, 1822(6): 875-84. |
 2015, Vol. 42
2015, Vol. 42


