哺乳动物基因组中蛋白质编码基因(protein coding gene,PCG)的比例不足2%,但剩下的巨 大的非蛋白编码区并非“垃圾DNA”,而是包含大 量转录调控元件和非编码RNA (noncoding RNA, ncRNA)基因。ncRNA通常是指不能翻译为蛋白 的RNA分子,包含短链ncRNA(如microRNA、 piRNA、siRNA等)和长链非ncRNA(lncRNA)。 目前发现或预测的人ncRNA已经超过了PCG,达到 4万以上,其中主要为lncRNA。lncRNA可在各个层 面调控基因表达,在细胞生长、发育、衰老、死亡 等过程中均扮演关键角色。 1 lncRNA概述 1.1 lncRNA的定义
lncRNA通常指一类相对分子质量大于200 nt, 且不能翻译为蛋白质的RNA分子。然而随着 研究的深入,发现该定义已经不能涵盖全部的 lncRNA。有学者建议将lncRNA定义为与传统类型 小RNA及结构RNA不同的,在初始转录或剪接后 水平调控基因的功能性RNA分子[ 1, 2 ]。 1.2 lncRNA的分类
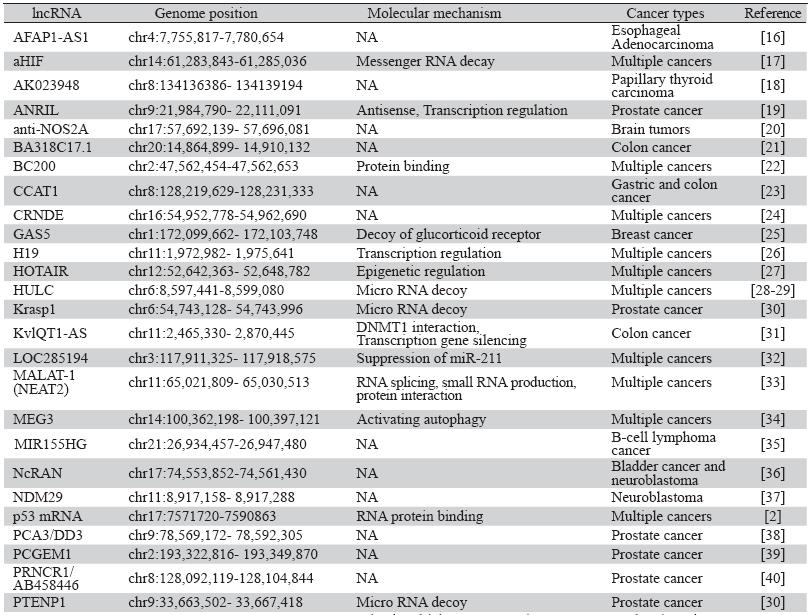
目前lncRNA的分类和命名比较混乱,缺乏统 一标准[ 3 ]。可根据来源将其分为:(1)由PCG的基 因结构中断形成新的lncRNA;(2)染色质重排产 生出一个新的含有多个外显子的lncRNA;(3)非 编码基因在复制过程中由于反转座作用导致基因重 叠而产生的lncRNA;(4)局部的复制子串联产生 的lncRNA;(5)基因中插入一个转座元件而产生 的lncRNA。可根据在基因组定位的不同将lncRNA 分为:(1)反义lncRNA;(2)内含子lncRNA; (3)基因间lncRNA;(4)启动子相关lncRNA; (5)非翻译区lncRNA等。目前已经有很多在线数 据库开始收录lncRNA的相关信息,见表 1。
|
|
表 1 lncRNA公共数据库 Table 1 lncRNA in public databases |
lncRNA参与了基因组印迹、X染色体失活、 染色体修饰以及端粒生物学等多种生命活动过 程,参与人类多种疾病的发生和发展,尤其与恶 性肿瘤的发生、发展关系密切。由于lncRNA种类 较多,功能及作用机制复杂,目前尚知之甚少, 但也发现lncRNA的功能存在一定的共同点,可以 在转录水平、转录后水平以及表观遗传水平(如 染色质重塑、组蛋白修饰)调控基因表达。根据 现有的研究结果,研究者提出了一些作用模型, 鉴于已有中文综述提及[ 15 ],本文不再展开。 2 肿瘤相关lncRNA
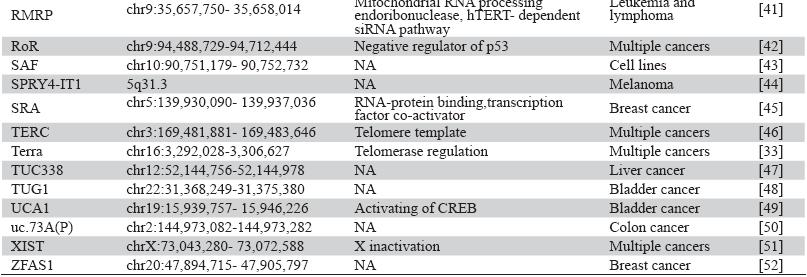
关于lncRNA的差异表达与肿瘤发生发展的 关系已经研究了很多年,然而,一直缺乏强有力 的直接证据。近年来随着肿瘤转录组表达谱的完 善,越来越多的证据揭示了lncRNA的功能,并发 现了大量的肿瘤相关lncRNA分子,见表 2。下面就 一些具有显著特点的肿瘤相关lncRNAs作一综述。
2.1 MALAT1这一大小约7.5 kb的lncRNA在人类多种肿瘤, 如肺癌、乳腺癌、胰腺癌、结肠癌以及前列腺癌 中异常高表达,其中尤以在非小细胞肺癌中的过 表达最为显著。研究已证实MALAT1的高水平表 达可促进肿瘤细胞转移。MALAT1的可能分子机 制包括剪接调节和基因表达调节。MALAT1可激 活转移相关基因的表达而促进肿瘤转移[ 33 ],还可 通过调节丝氨酸/精氨酸剪接因子的磷酸化水平使 mRNA前体的剪接发生改变[ 53 ],进而影响相应基因 的表达与功能。 2.2 HULC
HULC在肝癌和大肠癌肝转移中的表达显著增 高。HULC与乙肝病毒X蛋白HBx介导的肝癌发生 密切相关。HBx能够通过cAMP应答元件结合蛋白 (CREB)使HULC启动子激活[ 28 ]。此外,HULC 还可作为“分子诱饵”或是“miRNA海绵”在转录水 平下调miR-372的表达,导致其靶基因PRKACB水 平上调,激活CREB,CREB活化后与HULC启动 子结合从而促进HULC的转录[ 29 ]。 2.3 XIST
X染色体失活是哺乳动物表观遗传学调节的一 种经典模型,XIST是其主要调节因子。在雌性动 物细胞中,XIST可通过招募EZH2、RING-1等因 子到一条X染色体上,导致其失活,从而弥补了雌 雄性之间X相关基因数量的不平衡。乳腺癌中,可 存在两条活性X染色体。其共同机制是非活性X染 色体发生缺失或活性X染色体发生复制导致XIST 异常表达[ 51 ]。 2.4 HOTAIR
HOTAIR是第一个被证明可调控肿瘤转移的 lncRNA,在乳腺癌、肝细胞癌、喉鳞状细胞癌等 恶性肿瘤中表达显著上调,并与肿瘤的分期、转 移以及生存密切相关[ 27 ]。HOTAIR可募集PRC2复 合体并将其定位到HOXD位点,导致该位点上一大 小为40 kb的区域发生表达沉默,从而改变乳腺上 皮细胞的表型,使之类似于胚胎的纤维原细胞[ 54 ]。 HOTAIR还可能通过促进PTEN的甲基化导致其失 活进而促进肿瘤发生发展[ 55 ]。 2.5 GAS5
GAS5位于1q25,该区域在多种肿瘤中存在结 构异常。GAS5在缺乏营养或生长因子条件下大量 表达,扮演RNA Decoy角色,从而竞争抑制糖皮 质激素等类固醇激素介导的转录。GAS5在控制哺 乳动物细胞凋亡和细胞增殖过程中起重要作用, 可使乳腺癌细胞对凋亡诱导物更加敏感[ 25 ]。 2.6 H19
H19在脊椎动物胚胎发育过程中高表达,但出 生后不久即显著下调(除骨和软骨组织外)。H19 在多种肿瘤组织中异常高表达,可被癌基因c-Myc 直接活化,而抑癌基因p53则可下调其表达[ 26 ]。肝 脏中的H19与其作用靶点—血管生成素和成纤维细 胞生长因子之间相互作用,可改变与血管转移、 肿瘤浸润和细胞迁移相关基因的表达从而诱发肿 瘤。但也有证据表明H19可作为抑癌基因,如缺失 H19的小鼠较野生型更易发生肿瘤样息肉[ 56 ]。H19 同时兼有原癌基因和抑癌基因的作用,可能与其 特性相关,也可能与肿瘤的微环境有关[ 3 ]。 2.7 MEG3
这是第一个被发现具有肿瘤抑制功能的 lncRNA,其大小约1.6 kb。MEG3在人类很多正常 组织中表达,却在多种肿瘤组织中表达下调或丧 失,其机制包括基因缺失、启动子超甲基化等。 MEG3可以激活p53依赖或p53非依赖的信号通路抑 制肿瘤生成。由MEG3介导的p53的活化是依赖其 二级结构而不是其原始序列发挥作用的[ 34 ]。 2.8 UCA1
UCA1属于人内生性逆转录酶病毒H家族,在 绒膜绒毛、胚胎和胎儿膀胱组织中高表达。UCA1 在膀胱癌中表达上调,可促进膀胱癌细胞的增殖 和转移能力,并可通过CREB经由PI3K-AKT通路 调节细胞周期[ 49 ]。UCA1还可通过调控多个细胞周 期相关基因来改变细胞周期进程。 2.9 DD3
DD3基因位于9q21.22位点,目前尚未发现与 其同源的任何基因。现有资料表明DD3仅在前列 腺中特异性高表达,对前列腺癌的诊断价值要高 于目前常用的端粒酶逆转录酶,具有广阔的应用 前景[ 38 ]。 2.10 PTENP1
PTENP1是抑癌基因PTEN的假基因,与 其3 ’UTR高度相似,可竞争结合靶向PTEN的 miRNA(如miR-17、miR-21和miR-19等),从而解除 这些miRNA对PTEN的翻译抑制。PTENP1的发现 表明不同类型的ncRNA(miRNA和lncRNA)可共 同发挥基因调控功能[ 30 ]。 3 lncRNA在肿瘤诊断与治疗中的潜在应用
近年的研究显示,肿瘤的发生、发展、转移 与预后都与lncRNA相关。因此,lncRNA可作为 新的标志物用于肿瘤的诊断、预后和疗效监测。 例如,DD3用于前列腺癌诊断的敏感度和准确率 类似于PSA,且联合检测DD3和PSA可提高前列腺 癌的早期诊断率;检测尿沉渣UCA1用于膀胱癌的 诊断具有较高的特异性和敏感度;分析非小细胞 肺癌组织中MALAT1的含量可以早期预测肿瘤转 移;而HOTAIR是喉鳞状细胞癌、鼻咽癌的独立预 后因子[ 55 ]。
目前,将lncRNA用于肿瘤治疗干预靶点的研 究还处于起步阶段。尽管目前对lncRNA功能机制 所知有限,但已经为当前肿瘤药物的研发策略带来 很多提示,如合成可形成特定二级结构的RNA分 子,模拟lncRNA对基因的调控功能,抑制肿瘤的 增殖和转移。已经有部分lncRNA显示出作为肿瘤 干预靶点的巨大应用前景。例如,阻止HOTAIR与 PCR2复合物的反应,可限制乳腺癌细胞的转移潜 能。抑制GAS5表达不仅可诱导乳腺癌细胞的生长 停滞和凋亡,还可增加肿瘤对化疗药物的敏感度, 提示GAS5未来可能用于乳腺癌的预防和治疗[ 25 ]。 目前已经有一些靶向lncRNA的小分子化合物进入 了临床试验阶段。 4 展望
虽然lncRNA的研究已经进行了很多年,但 一直进展缓慢,总体上仍处于起步阶段,绝大多 数lncRNA的功能机制还不清楚。由于lncRNA种 类众多,结构多样,导致其作用机制复杂,目前 尚未建立成熟的研究技术体系,仅能对少数几类 lncRNA进行研究。此外,目前为止仍没有一个完 善的lncRNA数据库,仅建立了少数几个lncRNA转 基因模型。由于lncRNA广泛的生理功能和病理意 义,对lncRNA进行深入研究不仅会大大促进后基 因组时代对基因调控的理解,也必将对生物医学 的发展带来革命性的发展。
| [1] | Spizzo R, Almeida MI, Colombatti A, et al. Long non-coding RNAs and cancer: a new frontier of translational research?[J].Oncogene,2012,31(43):4577-87. |
| [2] | Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet,2009,10(3):155-9. |
| [3] | Gibb EA, Brown CJ, Lam WL. The functional role of long noncoding RNA in human carcinomas[J]. Mol Cancer,2011,10:38. |
| [4] | Erdmann VA, Szymanski M, Hochberg A, et al. Non-coding, mRNAlike RNAs database Y2K[J]. Nucleic Acids Res,2000,28(1):197-200. |
| [5] | Liu C, Bai B, Skogerb? G, et al. NONCODE: an integrated knowledge database of non-coding RNAs[J]. Nucleic Acids Res,2005,33(Database issue):D112-5. |
| [6] | Szymanski M, Erdmann VA, Barciszewski J. Noncoding RNAs database (ncRNAdb)[J]. Nucleic Acids Res,2007,35(Database issue):D162-4. |
| [7] | Mituyama T, Yamada K, Hattori E, et al. The Functional RNA Database 3.0: databases to support mining and annotation of functional RNAs[J]. Nucleic Acids Res,2009,37(Database issue):D89-92. |
| [8] | Zhang Y, Guan DG, Yang JH, et al. ncRNAimprint: a comprehensive database of mammalian imprinted noncoding RNAs[J]. RNA,2010,16(10):1889-901. |
| [9] | Dinger ME, Pang KC, Mercer TR, et al. NRED: a database of long noncoding RNA expression[J]. Nucleic Acids Res,2009,37(Database issue):D122-6. |
| [10] | Amaral PP, Clark MB, Gascoigne DK, et al. lncRNAdb: a reference database for long noncoding RNAs[J]. Nucleic Acids Res,2011,39(Database issue):D146-51. |
| [11] | Burge SW, Daub J, Eberhardt R,et al. Rfam 11.0: 10 years of RNA families[J]. Nucleic Acids Res 2013, 41(Database issue):D226-32. |
| [12] | Bejerano G, Pheasant M, Makunin I,et al. Ultraconserved elements in the human genome[J]. Science,2004,304(5675):1321-5. |
| [13] | Volders PJ, Helsens K, Wang X, et al. LNCipedia: a database for annotated human lncRNA transcript sequences and structures[J]. Nucleic Acids Res,2013,41(Database issue):D246-51. |
| [14] | Wu T, Wang J, Liu C,et al. NPInter: the noncoding RNAs and protein related biomacromolecules interaction database[J]. Nucleic Acids Res,2006,34(Database issue):D150-2. |
| [15] | Gao Y, Hui N, Liu S. Long Non-coding RNA:research progress[J]. Di Er Jun Yi Da Xue Xue Bao,2011,32(7):790-4.[高原, 惠宁, 刘善荣. 长链非编码RNA的研究进展[J]. 第二军医大学学报,2011,32(7):790-4.] |
| [16] | Wu W, Bhagat TD, Yang X, et al. Hypomethylation of noncoding DNA regions and overexpression of the long noncoding RNA, AFAP1-AS1, in barrett’s esophagus and esophageal adenocarcinoma[J]. Gastroenterology,2013,144(5):956-66.e4. |
| [17] | Rossignol F, Vaché C, Clottes E. Natural antisense transcripts of hypoxia-inducible factor 1alpha are detected in different normal and tumour human tissues[J]. Gene,2002,299(1-2):135-40. |
| [18] | He H, Nagy R, Liyanarachchi S, et al. A susceptibility locus for papillary thyroid carcinoma on chromosome 8q24[J]. Cancer Res,2009,69(2):625-31. |
| [19] | Kotake Y, Nakagawa T, Kitagawa K, et al. Long non-coding RNA ANRIL is required for the PRC2 recruitment to and silencing of p15(INK4B) tumor suppressor gene[J]. Oncogene,2011,30(16): 1956-62. |
| [20] | Korneev SA, Korneeva EI, Lagarkova MA, et al. Novel noncoding antisense RNA transcribed from human anti-NOS2A locus is differentially regulated during neuronal differentiation of embryonic stem cells[J]. RNA,2008,14(10):2030-7. |
| [21] | Davison EJ, Tarpey PS, Fiegler H, et al. Deletion at chromosome band 20p12.1 in colorectal cancer revealed by high resolution array comparative genomic hybridization[J]. Genes Chromosomes Cancer,2005,44(4):384-91. |
| [22] | Chen W, B?cker W, Brosius J, et al. Expression of neural BC200 RNA in human tumours[J]. J Pathol,1997,183(3):345-51. |
| [23] | Yang F, Xue X, Bi J, et al. Long noncoding RNA CCAT1, which could be activated by c-Myc, promotes the progression of gastric carcinoma[J]. J Cancer Res Clin Oncol,2013,139(3):437-45. |
| [24] | Ellis BC, Molloy PL, Graham LD. CRNDE: A Long Non-Coding RNA Involved in CanceR, Neurobiology, and DEvelopment[J]. Front Genet,2012,3:270. |
| [25] | Mourtada-Maarabouni M, Pickard MR, Hedge VL, et al. GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer[J]. Oncogene,2009,28(2):195-208. |
| [26] | Dugimont T, Montpellier C, Adriaenssens E, et al. The H19 TATA-less promoter is efficiently repressed by wild-type tumor suppressor gene product p53[J]. Oncogene,1998,16(18):2395-401. |
| [27] | Gupta RA, Shah N, Wang KC,et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis[J]. Nature,2010,464(7291):1071-6. |
| [28] | Du Y, Kong G, You X, et al. Elevation of highly up-regulated in liver cancer (HULC) by hepatitis B virus X protein promotes hepatoma cell proliferation via down-regulating p18[J]. J Biol Chem,2012,287(31):26302-11. |
| [29] | Wang J, Liu X, Wu H, et al. CREB up-regulates long non-coding RNA, HULC expression through interaction with microRNA-372 in liver cancer[J]. Nucleic Acids Res,2010,38(16):5366-83. |
| [30] | Poliseno L, Salmena L, Zhang J, et al. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology[J]. Nature,2010,465(7301):1033-8. |
| [31] | Tanaka K, Shiota G, Meguro M, et al. Loss of imprinting of long QT intronic transcript 1 in colorectal cancer[J]. Oncology,2001,60(3):268-73. |
| [32] | Liu Q,Huang J,Zhou N,et al.LncRNA loc285194 is a p53-regulated tumor suppressor[J]. Nucleic Acids Res,2013,41(9):4976-87. |
| [33] | Gutschner T, H?mmerle M, Eissmann M, et al. The non-coding RNA MALAT1 is a critical regulator of the metastasis phenotype of lung cancer cells[J]. Cancer Res,2013,73(3):1180-9. |
| [34] | Zhang X, Rice K, Wang Y, et al. Maternally expressed gene 3 (MEG3) noncoding ribonucleic acid: isoform structure, expression, and functions[J]. Endocrinology,2010,151(3):939-47. |
| [35] | Eis PS, Tam W, Sun L,et al. Accumulation of miR-155 and BIC RNA in human B cell lymphomas[J]. Proc Natl Acad Sci U S A,2005,102(10):3627-32. |
| [36] | Zhu Y, Yu M, Li Z,et al. ncRAN, a newly identified long noncoding RNA, enhances human bladder tumor growth, invasion, and survival[J].Urology,2011,77(2):510.e1-5. |
| [37] | Castelnuovo M, Massone S, Tasso R, et al. An Alu-like RNA promotes cell differentiation and reduces malignancy of human neuroblastoma cells[J]. FASEB J,2010,24(10):4033-46. |
| [38] | de Kok JB, Verhaegh GW, Roelofs RW, et al. DD3(PCA3), a very sensitive and specific marker to detect prostate tumors[J]. Cancer Res,2002,62(9):2695-8. |
| [39] | Ifere GO, Ananaba GA. Prostate cancer gene expression marker 1 (PCGEM1): a patented prostate- specifi c non-coding gene and regulator of prostate cancer progression[J]. Recent Pat DNA Gene Seq,2009,3(3):151-63. |
| [40] | Chung S,Nakagawa H,Uemura M,et al. Association of a novel long non-coding RNA in 8q24 with prostate cancer susceptibility[J]. Cancer Sci,2011,102(1):245-52. |
| [41] | Maida Y,Yasukawa M,Furuuchi M,et al. An RNA-dependent RNA polymerase formed by TERT and the RMRP RNA[J].Nature, 2009,461(7261):230-5. |
| [42] | Zhang A, Zhou N, Huang J, et al. The human long non-coding RNA-RoR is a p53 repressor in response to DNA damage[J]. Cell Res,2013,23(3):340-50. |
| [43] | Yan MD,Hong CC,Lai GM,et al. Identifi cation and characterization of a novel gene Saf transcribed from the opposite strand of Fas[J]. Hum Mol Genet,2005,14(11):1465-74. |
| [44] | Khaitan D, Dinger ME, Mazar J, et al. The melanoma-upregulated long noncoding RNA SPRY4-IT1 modulates apoptosis and invasion[J]. Cancer Res,2011,71(11):3852-62. |
| [45] | Lanz RB, McKenna NJ, Onate SA, et al. A steroid receptor coactivator, SRA, functions as an RNA and is present in an SRC-1 complex[J]. Cell,1999,97(1):17-27. |
| [46] | Cao Y, Bryan TM, Reddel RR. Increased copy number of the TERT and TERC telomerase subunit genes in cancer cells[J]. Cancer Sci,2008,99(6):1092-9. |
| [47] | Braconi C, Valeri N, Kogure T, et al. Expression and functional role of a transcribed noncoding RNA with an ultraconserved element in hepatocellular carcinoma[J]. Proc Natl Acad Sci U S A,2011,108(2):786-91. |
| [48] | Han Y, Liu Y, Gui Y, et al. Long intergenic non-coding RNA TUG1 is overexpressed in urothelial carcinoma of the bladder[J]. J Surg Oncol,2013,107(5):555-9. |
| [49] | Yang C,Li X,Wang Y,et al. Long non-coding RNA UCA1 regulated cell cycle distribution via CREB through PI3-K dependent pathway in bladder carcinoma cells[J]. Gene,2012,496(1):8-16. |
| [50] | Calin GA, Liu CG, Ferracin M, et al. Ultraconserved regions encoding ncRNAs are altered in human leukemias and carcinomas[J]. Cancer Cell,2007,12(3):215-29. |
| [51] | Sirchia SM, Tabano S, Monti L, et al. Misbehaviour of XIST RNA in breast cancer cells[J]. PLoS One,2009,4(5):e5559. |
| [52] | Askarian-Amiri ME, Crawford J, French JD, et al. SNORDhost RNA Zfas1 is a regulator of mammary development and a potential marker for breast cancer[J]. RNA,2011,17(5):878-91. |
| [53] | Tripathi V, Ellis JD, Shen Z, et al. The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation[J]. Mol Cell,2010,39(6):925-38. |
| [54] | Tsai MC, Manor O, Wan Y,et al. Long noncoding RNA as modular scaffold of histone modifi cation complexes[J]. Science,2010,329 (5992):689-93. |
| [55] | Li D, Feng J, Wu T, et al. Long intergenic noncoding RNA HOTAIR is overexpressed and regulates PTEN methylation in laryngeal squamous cell carcinoma[J]. Am J Pathol,2013,182(1):64-70. |
| [56] | Yoshimizu T, Miroglio A, Ripoche MA, et al. The H19 locus acts in vivo as a tumor suppressor[J]. Proc Natl Acad Sci U S A,2008,105(34):12417-22. |
 2014, Vol.41
2014, Vol.41