2.唐山市工人医院病理科
2.Department of Pathology, Tangshan Gongren Hospital
微小RNA(miRNA)是新发现的长度为19~24 个核苷酸的非编码小分子单链RNA,可通过调控 靶基因的表达,在细胞分化、细胞周期及凋亡过 程中发挥重要作用。近来,miRNA与肿瘤的相关 性备受关注。研究发现,miRNA兼有类似癌基因 及抑癌基因的作用,在肿瘤的发生发展中起重要 作用[1]。对肿瘤及正常组织中miRNA的差异表达 谱研究,有助于筛选特定功能的miRNA。本研究 采用miRNA芯片技术筛选直肠癌组织与正常组织 差异表达的miRNA,并对部分差异miRNA进行验 证,报道如下。 1 资料与方法 1.1 标本采集
随机选取2008年1月—2010年1月间在河北医 科大学第一医院普外科行手术治疗的直肠癌患者 手术标本18例,病理诊断均为直肠腺癌,所有患 者术前均未接受放疗、化疗。取癌组织及上切缘 正常黏膜(术后病理证实无癌组织侵犯),所取 标本离体时间均小于10 min,将组织块剪至约1 cm3 大小,DEPC-PBS冲洗后迅速置于液氮中,后转至-80℃保存。 1.2 试剂
DEPC,Trizol 试剂购于美国 Invitrogen公 司,RNeasy mini kit RNA 试剂盒购于Qiagen 公 司,miRNA array相关试剂为美国LC science公 司产品。 Prime Script TM RT reagent Kit反转 录试剂盒和荧光定量PCR检测试剂盒购自美国 Ambion 公司,Taqman 引物为life technology 公 司产品(RNU6B:001093;miR-187:000487; miR-224:000599)。 1.3 试验方法 1.3.1 总RNA抽提
每100 mg组织样品加入1 ml 的Trizol 试剂,匀浆器匀浆。所加样品体积不超过 Trizol 试剂体积的10% 。按RNeasy mini kit RNA试 剂盒说明书纯化总RNA。 1.3.2 总RNA质量检测
使用NanoDrop®ND- 1000测定RNA在260、280和230 nm的OD值,计算 浓度并评估纯度。进行变性琼脂糖凝胶电泳,以 检测RNA纯度及完整性。 1.3.3 ParafloTM microRNA微阵列芯片分析
ParafloTM microRNA微阵列芯片分析由美国LC Sciences公 司完成。用YM-100离心过滤器(Millipore)分 离10 μg总RNA获得小RNAs。小RNA的3' 端用T4 RNA连接酶标记荧光染料Cy3和Cy5。用μParaflo 微流体芯片检测,微阵列芯片上的每个探针至 少重复3次,miRNA探针序列信息来自于Sanger miRBase Release 17.0 版本数据库。每个微阵列 实验都含有 16组质控探针用于质量控制。选择组 织内源的5S rRNA 作为miRNA芯片的内源阳性对 照;同时设立阴性对照和空白对照。 1.3.4 芯片扫描及数据处理
利用激光扫描仪 (Gene Pix 4000B,Molecular Device)采集杂 交图像并使用Array-Pro图像分析软件(Media Cybernetics)进行图像数字化转换。数据分析首先 是减去背景值,然后使用LOWESS过滤进行信号 归一化,将数据进行统计学处理,筛选出差异表 达的miRNA(P<0.05)。 1.3.5 实时定量RT-PCR为核对芯片的准确性
选 取有代表性的差异表达的miRNA(hsa-miR-187* 和miR-224)进行实时定量RT-PCR,以U6作为内 参。将1 μg 总RNA反转录为cDNA,以cDNA为模 板进行PCR 扩增,95℃ 变性10 min,95℃ 15 s; 60℃ 1 min;共40个循环。 2 结果 2.1 总RNA提取的完整性及浓度
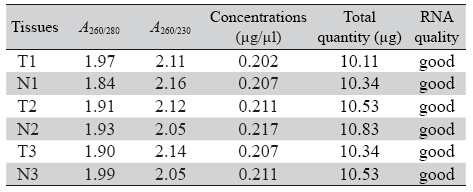
miRNA表达谱芯片试验要求评价标准为:RNA纯度:A260/280在1.8~2.1之间,RNA总量≥3 μg,总 RNA完整性:经琼脂糖凝胶电泳检测,RNA样品 电泳28S、18S rRNA 条带清晰且28S:18S rRNA 条 带亮度大于或接近2:1。结果显示,全部标本RNA 完整性及浓度符合实验要求。组织总RNA提取的 完整性及浓度,见表 1。琼脂糖凝胶电泳检测总 RNA 完整性结果,见图 1。
|
|
表 1 组织总RNA 提取的完整性及浓度 Table 1 Details of total RNA isolated from rectal tissues |
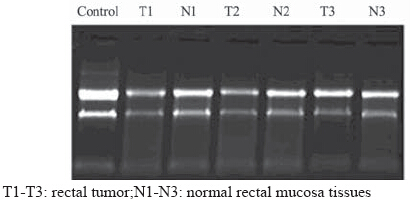 |
图 1 琼脂糖凝胶电泳检测总RNA完整性 Figure 1 Total RNA integrity detected by agarose gel electrophoresis |
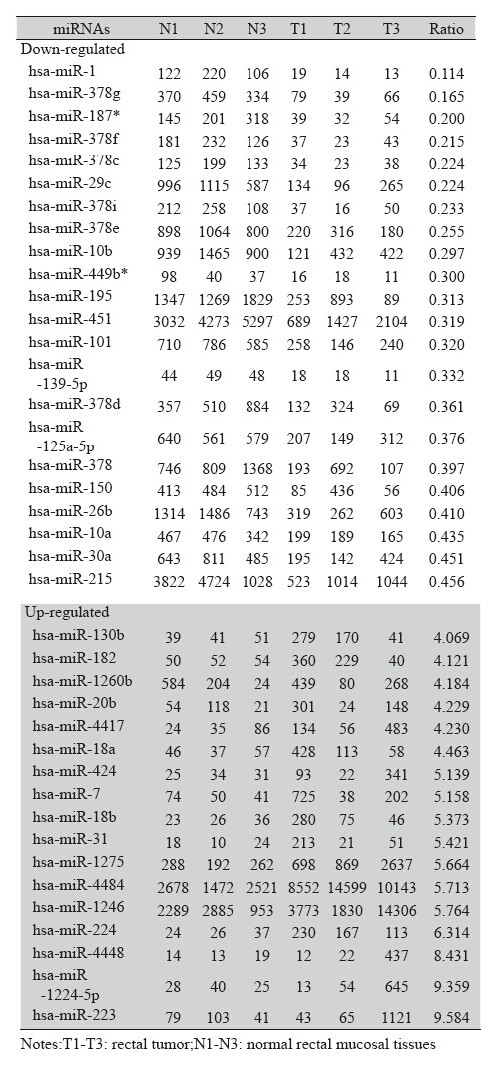
将抽提所得的直肠癌及正常黏膜总RNA分 别进行miRNA芯片分析,结果显示直肠癌组织 miRNA表达谱与正常直肠黏膜有明显差异。聚 类分析可以将直肠腺瘤组织与正常黏膜组织明确 区分开,见图 2。为了找出正常直肠黏膜与直肠 癌组织miRNA表达的差异,将芯片中的数据经归 一化后进行统计分析,直肠癌组织与正常直肠黏 膜杂交信号强度的比值(T/N)>2为上调,<0.5 为下调。共筛选出70个直肠癌相关的差异表达 miRNA,其中33个表达上调,37个表达下调。其 中T/N<0.5的有22个,T/N >4的有17个,见表 2。
 |
图 2 直肠癌与正常黏膜组织miRNAs 差异表达分层聚类图 Figure 2 Hierarchical clustering map of differential expressions of miRNAs between rectal tumor and normal rectal mucosal tissues |
|
|
表 2 直肠癌组织与正常直肠黏膜表达差异的miRNAs Table 2 Differential expressions of miRNAs in rectal tumor and rectal mucosal tissues |
根据ratio比值,我们分别选取hsa-miR-187*以 及hsa-miR-224进行实时定量RT-PCR,计算公式: 相对表达量=2-△△CT,-△CT=CT待测miRNA-CTU6。结 果如图所示:miR-187*在直肠癌组织中的表达较 正常直肠黏膜明显降低,见图 3A,而miR-224在癌 组织中的表达明显增高(P均<0.001),见图 3B, 与miRNA芯片结果,见图 3C,具有一致性。
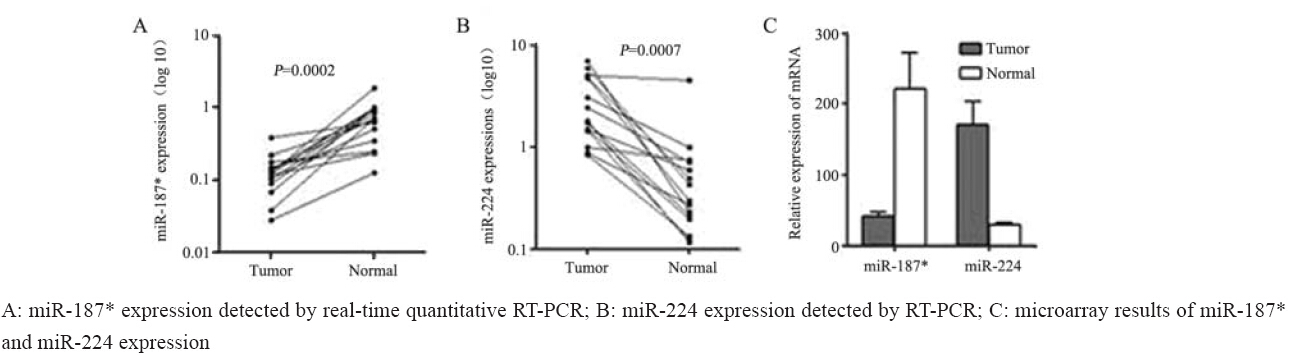 |
图 3 RT-PCR 和基因芯片检测miR-187* 和miR-224的表达 Figure 3 Comparision of miR-187* and miR-224 expressions detected by real-time quantitative RT-PCR and microarray analysis |
直肠癌是我国常见的恶性肿瘤,全国范围内其发病率高于结肠癌,直肠癌早期预后较好,而进展 期预后较差[2]。研究发现,miRNA与肿瘤关系密切, 可通过抑制转录后水平的蛋白翻译调节多种生物学 信号通路,参与调控肿瘤的发生和发展过程[3, 4, 5]。
由于肿瘤组织中存在特定的miRNA表达谱, 可能在肿瘤的诊断及治疗方面发挥潜在的作用。 为了得到直肠癌差异表达的miRNA,本实验选 择了3例进展期直肠腺癌患者(均伴有淋巴结转 移),采用 microRNA Arrays技术分析正常直肠粘 膜、直肠腺癌组织中差异表达的miRNAs。该芯片含有1719条成熟的miRNAs探针,与成熟miRNA 完全互补,能特异性地检测出成熟的miRNA并能 很好地区分高度同源的miRNA分子,该平台还具 有特异性高、敏感度高等特点。经研究发现,直 肠腺癌与正常直肠黏膜相比,多个miRNA存在差 异性表达。对比文献报道,发现部分miRNA表达 变化与既往报道相吻合,上调的包括miR-182、 miR-17、miR-106a、miR-18a、miR-31、miR-224 等。下调的miRNA包括为miR-1、miR-29c、 miR-101、miR-139-5p、miR-150等。同时我们还筛 选出一些新的可能与直肠癌发展有关的miRNAs。
为了进一步验证芯片的准确性,我们分别挑 选了miR-187*和miR-224进行荧光定量RT-PCR 检测,结果证实与正常组织相比,直肠癌中hsamiR- 224的表达上调,hsa-miR-187*表达下调。 miR-187*(miR-187-5p)是miR-187家族的一员, 已有研究证实,卵巢癌和乳腺癌中miR-187呈高表 达且乳腺癌中miR-187的高表达和患者的不良预后 有关[6, 7],而前列腺癌和胰腺癌细胞中miR-187低 表达[8, 9]。目前关于miR-187*在肿瘤中的研究鲜有 报道,Liu等[10]发现miR-187*在胃癌患者中表达水 平明显升高,Ho等[11]研究证实,毛细胞型星形细 胞瘤标本中miR-187*的表达水平明显低于正常组 织。然而未见在直肠癌组织中的相关研究。本研 究证实,miR-187*在直肠癌组织中表达下调,可 能发挥类似抑癌基因的作用,但具体的作用机制 尚待后续实验研究。
本实验中得到的另外一种差异表达miRNA— miR-224,在直肠癌组织中表达明显上调。研究证实 miR-224在多种肿瘤组织和细胞中表达上调,如肝 癌[12]、子宫颈癌、胶质瘤等,且miR-224的高表达和 宫颈癌及胶质瘤患者的预后不良有关[13, 14]。然而有学者发现,在乳腺粘液癌患者及前列腺癌中miR-224 的表达下调[15, 16]。许多芯片筛选研究结果发现,结 直肠癌组织中miR-224表达上调[17],与我们芯片筛 选结果一致,如Vega等[18]发现miR-224在结直肠癌 三期患者肿瘤组织中表达上调,Olaru等[19]发现炎 症性肠病相关CRC患者肿瘤组织中miR-224表达上 调,且可能通过靶向调节p21蛋白发挥作用。肝细 胞癌中研究发现miR-224可以通过降低凋亡抑制 因子-5(apoptosis inhibitor 5,API-5)而参与细胞凋 亡[20],同时参与肝癌细胞的侵袭和转移相关[21]。 关于miR-224在直肠癌中的作用及具体机制及参与 调控的细胞通路尚需进一步研究。
总之,我们应用芯片技术筛选出直肠癌特异性 miRNA表达谱,同时选取的均为Dukes C期或D期直 肠癌样本,相对于混合不同病理分期的实验样本, 增加了鉴定异常表达 miRNAs 的敏感度。直肠癌发 生过程中存在特异性miRNA改变,但其靶基因及具 体作用机制尚待阐明。相信随着各方面研究的深入, miRNA将在直肠癌的早期诊治方面发挥重要作用。
| [1] | He X, Dong Y, Wu CW, et al. MicroRNA-218 inhibits cell cycle progression and promotes apoptosis in colon cancer by downregulating oncogene BMI-1 polycomb ring finger oncogene[J]. Mol Med, 2013, 18:1491-8. |
| [2] | Liu YL, Zhao FL, Wang XS. Analysis of clinicopathologic features and prognosis in 2414 patients with rectal cancer[J]. Zhonghua Wei Chang Wai Ke Za Zhi, 2010,13(12): 913-6. [刘彦 龙, 赵发林, 王锡山. 2414例直肠癌患者的临床病理特征及预后 分析[J].中华胃肠外科,2010,13(12):913-6.] |
| [3] | Chen X, Guo X, Zhang H, et al. Role of miR-143 targeting KRAS in colorectal tumorigenesis[J]. Oncogene, 2009, 28(10): 1385-92. |
| [4] | Liu L, Chen L, Xu Y, et al. microRNA-195 promotes apoptosis and suppresses tumorigenicity of human colorectal cancer cells[J]. Biochem Biophys Res Commun, 2010, 400(2): 236-40. |
| [5] | Liang L, Li X, Zhang X, et al. microRNA-137, a HMGA1 target, suppresses colorectal cancer cell invasion and metastasis in mice by directly targeting FMNL2[J]. Gastroenterology, 2013,144(3): 624-35.e4. |
| [6] | Chao A, Lin CY, Lee YS, et al. Regulation of ovarian cancer progression by microRNA-187 through targeting Disabled homolog-2[J]. Oncogene,2012, 31(6):764-75. |
| [7] | Mulrane L, Madden SF, Brennan DJ, et al. miR-187 is an independent prognostic factor in breast cancer and confers increased invasive potential in vitro[J]. Clin Cancer Res, 2012, 18(24):6702-13. |
| [8] | Fuse M, Kojima S, Enokida H,et al. Tumor suppressive microRNAs (miR-222 and miR-31) regulate molecular pathways based on microRNA expression signature in prostate cancer[J]. J Hum Genet, 2012, 57(11):691-9. |
| [9] | Schultz NA, Andersen KK, Roslind A, et al. Prognostic microRNAs in cancer tissue from patients operated for pancreatic cancer--fi ve microRNAs in a prognostic index[J]. World J Surg, 2012, 36(11):2699-707. |
| [10] | Liu H, Zhu L, Liu B, et al. Genome-wide microRNA profiles identify miR-378 as a serum biomarker for early detection of gastric cancer[J]. Cancer Lett, 2012, 316(2):196-203. |
| [11] | Ho CY, Bar E, Giannini C,et al. MicroRNA profi ling in pediatric pilocytic astrocytoma reveals biologically relevant targets, including PBX3, NFIB, and METAP2[J]. Neuro Oncol, 2013, 15(1):69-82. |
| [12] | Zhang Y,Takahashi S,Tasaka A,et al.Involvement of microRNA-224 in cell proliferation, migration, invasion, and antiapoptosis in hepatocellular carcinoma[J]. J Gastroenterol Hepatol, 2013, 28(3):565-75. |
| [13] | Shen SN, Wang LF, Jia YF, et al. Upregulation of microRNA-224 is associated with aggressive progression and poor prognosis in human cervical cancer[J]. Diagn Pathol,2013,8:69. |
| [14] | Lu S, Wang S, Geng S, et al. Upregulation of microRNA-224 confers a poor prognosis in glioma patients[J]. Clin Transl Oncol,2013,15(7):569-74. |
| [15] | Zhou F, Li S, Meng HM,et al. MicroRNA and histopathological characterization of pure mucinous breast carcinoma[J]. Cancer Biol Med,2013,10(1):22-7. |
| [16] | Mavridis K, Stravodimos K, Scorilas A. Downregulation and prognostic performance of microRNA 224 expression in prostate cancer[J]. Clin Chem, 2013, 59(1):261-9. |
| [17] | Fu J, Tang W, Du P, et al. Identifying microRNA-mRNA regulatory network in colorectal cancer by a combination of expression profile and bioinformatics analysis[J]. BMC Syst Biol,2012,6:68. |
| [18] | Vega AB, Pericay C, Moya I, et al. microRNA expression profi le in stage III colorectal cancer: circulating miR-18a and miR-29a as promising biomarkers[J]. Oncol Rep,2013,30(1):320-6. |
| [19] | Olaru AV, Yamanaka S, Vazquez C, et al. MicroRNA-224 negatively regulates p21 expression during late neoplastic progression in inflammatory bowel disease[J]. Inflamm Bowel Dis,2013,19(3):471-80. |
| [20] | Wang Y, Lee AT, Ma JZ, et al. Profi ling microRNA expression in hepatocellular carcinoma reveals microRNA-224 up-regulation and apoptosis inhibitor-5 as a microRNA-224-specifi c target[J]. J Biol Chem,2008, 283(19):13205-15. |
| [21] | Li Q, Wang G, Shan JL, et al. MicroRNA-224 is upregulated in HepG2 cells and involved in cellular migration and invasion[J]. J Gastroenterol Hepatol,2010, 25(1):164-71. |
 2014, Vol.41
2014, Vol.41