2. 深圳市宝安区妇幼保健院乳腺科
2. Department of Breast Surgery, Bao’an Maternal and Child Health Hospital
乳腺癌是当今全球女性发病率位居第一的恶 性肿瘤,约占女性恶性肿瘤的23%[1]。乳腺癌的发 生发展是环境与遗传两因素共同作用的结果,其 中CpG岛启动子甲基化所致的抑癌基因失活被认 为是主要途径之一[2]。近年来大量的研究表明,视 黄醇受体β(retinoic acid receptor β,RARβ)基因作 用于恶性肿瘤细胞系的反转录过程,抑制肿瘤细 胞的增殖;如果失去了RARβ基因的调节作用就会 诱发恶性肿瘤的发生[3]。因此,RARβ作为乳腺癌 抑制因子备受关注,其甲基化与乳腺癌的发生发 展具有重大的关系[4]。然而,多数研究存在样本量 小或随访资料不全的缺点。本研究拟检测192例乳 腺癌组织中RARβ基因甲基化情况,结合临床病理 及随访资料,探讨RARβ基因甲基化与乳腺癌临床 病理参数及预后的关系。 1 资料与方法 1.1 资料
收集澳大利亚查尔斯爵士盖尔德纳医院手术 并病理确诊的未经化疗和放疗的原发性浸润性乳 腺癌及相应癌旁组织192例,平均年龄60 (18~93) 岁,随访时间(2~78) 月,平均51月。其中乳腺浸 润性导管癌共160例,浸润性小叶癌10例,浸润性 髓样癌9例,浸润性黏液癌1例;高-中分化腺癌96 例,低分化腺癌61例;有淋巴结转移者71例。39 例淋巴结转移情况、35例分化级别、23例组织学 分类、12例肿瘤组织学类型、15例肿瘤大小、5例 雌激素受体(ER)、孕激素受体(PR)表达情 况、77例倍数性及21例HER2表达情况数据缺乏, 具体资料见 表1。另取40例良性乳腺肿瘤患者的瘤 旁正常乳腺组织作为对照组。经西澳大利亚人类 研究伦理委员会批准研究。
|
|
表1 RARβ基因甲基化与乳腺癌临床病理的关系[例数(%)] Table 1 Relationship between RARβgene promoter methylation and clinicopathological features of breast cancer[n(%)] |
用亚硫酸氢钠修饰 DNA,MSP检测基因甲基化状态[5, 6]。1~2 μg DNA用 15 μl水溶解后加入1.7 μl 2M NaOH,混匀。37℃水 浴15 min。加入9 μl对苯二酚(1 mg/ml)和156 μl新 鲜制备的亚硫酸氢钠溶液(380 mg/ml),混匀; 最 后加滴50 μl矿物油,55℃水浴16 h。重亚硫酸盐修 饰后的DNA采用QIAquick PCR纯化试剂盒进行纯 化(Qiagen公司),具体步骤按操作说明进行。经50 μl 10 mM Tris-HCl (pH=8.0)洗涤,和5.5 μl 3M NaOH 脱磺基后,混匀室温下孵育15 min。然后再加入6 μl 3M醋酸钠(pH=5.2)、1 μl糖原(20 mg/ml)和180 μl 无水冰乙醇进行沉淀。随后将样品离心30 min后, 放置在-80℃30 min。小心吸去上清液,让管子在室 温下晾干,之后再加入20 ml Milli-Q水溶解。这种 亚硫酸氢钠修饰后的DNA随即直接用于PCR扩增。 1.2.2 引物设计
RARβ甲基化上游引物:5’-CG AGCGGGCGTAGGCGGAATATC-3’,下游引物: 5’-CAAACAACGAACGCACAAACCGACG-3’,扩 增产物为200 bp,退火温度为67℃,MgCl2浓度为2.5 mM;RARβ非甲基化上游引物:5’-GGTGAGTGGG TGTAGGTGGAATATT-3’,下游引物:5’-AACAAAC AACAAACACACAAACCAACAT-3’,扩增产物为 200 bp,退火温度为67℃,镁离子浓度为2.5 mM。 1.2.3 甲基化特异性PCR检测
Master mix 18 μl, 包括:修饰后的DNA 2 μl,1×聚合缓冲液,1×Q 液,2.5 mM MgCl2,上下游引物各0.4 μM,和Taq 酶0.1 μl (Qiagen公司)。PCR扩增条件::94℃ 预变性5 min,热循环94℃变性30 s,67℃退火45 s ,72℃延伸30 s,RARβ基因非甲基化循环35周 期,RAR基因甲基化循环38周期后,最后72℃延 伸7 min。用经SSS1酶处理的和未处理的正常人外 周血DNA作为甲基化及非甲基化的阳性对照。最 后,用2.0%的琼脂糖凝胶电泳分析PCR产物,如甲 基化引物扩增有条带则说明该样本发生了甲基化。 1.3 统计学方法
所有实验数据,均使用SPSS 17.0软件进行统计学 处理分析。采用χ2检验分析RARβ基因甲基化和乳腺 癌临床病理特点的关联性,Kaplan-Meier分析用来评 估累积生存率,而差异使用Log rank检验进行了评估。 2 结 果 2.1 RARβ基因的甲基化检测结果
40例正常乳腺组织中无一例检测到RARβ基因 甲基化,192例乳腺癌组织中50例有RARβ基因甲基 化(26%),差异有统计学意义(P<0.05)。运用MSP 技术检测乳腺癌RARβ基因甲基化的结果,见图 1。
 |
图 1 MSP分析法检测RARβ基因甲基化情况 Figure 1 RMSP analysis of RARβgene methylation in primary breast carcinomas |
χ2检验统计结果显示,RARβ基因甲基化率与 患者年龄、PR表达情况、倍数性及TP53突变无明 显相关性(P>0.05)。淋巴结转移、ER阴性及HER2 过度表达的癌组织中RARβ基因甲基化率增高,但 无统计学意义。直径>20 mm、Ⅲ期乳腺癌及浸润 性非导管型乳腺癌的RARβ基因甲基化率明显增高 (P<0.05)。在浸润性非导管型乳腺癌类型中RARβ 基因甲基化率最高(50%),见表1。RARβ基因 甲基化的肿瘤PR平均值(x±s)为(154±262) fmol/μg,高于无RARβ基因甲基化肿瘤PR平均值 (91±154)fmol/μg(P=0.04)。 2.3 RARβ基因甲基化与预后的关系
在本组乳腺癌患者,一些传统指标,如淋巴结 转移(P<0.001)、肿瘤大小(P<0.001)、组织 学分级(P<0.001)、TP53突变(P=0.002),均 与预后显著相关。RARβ基因甲基化与预后亦显示 类似的结果,生命曲线分析显示RARβ基因甲基化 的乳腺癌患者平均生存65.53(59.21~71.85)月,低 于无RARβ基因甲基化患者的69.06(65.84~72.28) 月,然而经Log rank检验,两组生存率曲线的差别 无统计学意义(χ2=1.23,P=0.268),见图 2A。
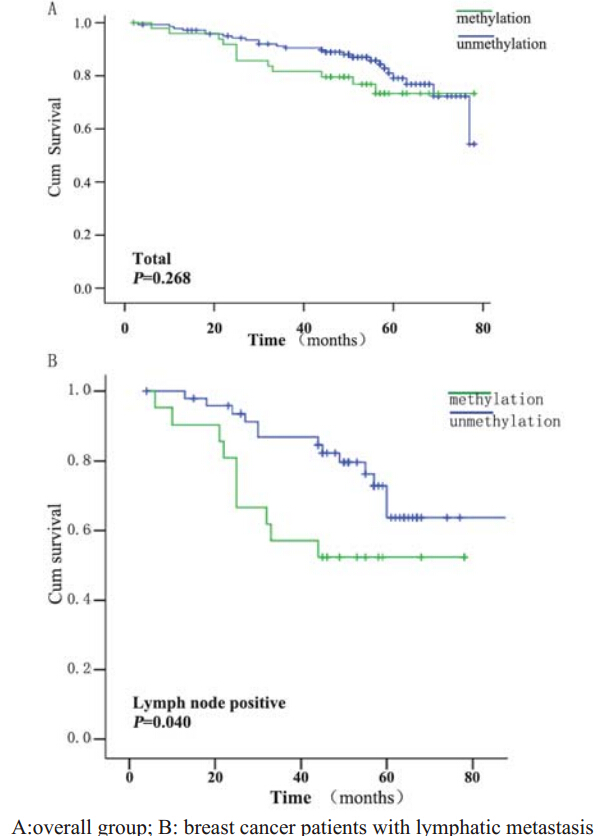 |
图 2 乳腺癌患者RARβ基因有无甲基化Kaplan–Meier分析生存率曲线图 Figure 2 Kaplan-Meier survival analysis for breast cancer patients with and without RARβmethylation |
在淋巴结转移的乳腺癌患者中,RARβ基因甲基化的患者平均生存52.43(40.56~64.30)月,明显低 于RARβ基因无甲基化的患者的73.13(65.12~81.14) 月,经Log rank检验,两组生存率曲线的差别有统计 学意义(χ2=4.230,P=0.040),见图 2B。另外,在肿 瘤直径>20 mm(χ2=3.353,P=0.067)和ER受体阳性 (χ2=3.149,P=0.076)的乳腺癌,RARβ基因甲基化 的患者均显示较差的预后结果,但无统计学意义。 3 讨论
本研究的目的是为了检测RARβ基因的甲基化 是否与乳腺癌的某些特异性表型包括预后相关联。 在大肠癌首次提出这种DNA甲基化特异性表型的发 现,被称为CpG岛甲基化表型(CIMP+)的一类大 肠癌[7]。那么,在乳腺癌是否也存在这种表型呢?
本实验用于检测RARβ基因甲基化的乳腺癌标 本的临床病理资料完整,一些传统的预后指标, 如淋巴结转移、组织学分级、肿瘤大小和TP53突 变,均具有统计学意义[8]。实验标本为新鲜冰冻的 乳腺癌组织,应用甲基化特异性PCR检测甲基化率 可达99.4% (1 343/1 351)。据文献报道,乳腺癌的 单个基因的甲基化率变化很大,可能与DNA的质 量、PCR循环周期和扩增条件有关。而且在不同基 因间甲基化特异性PCR的敏感度因PCR效率及其产 物的大小不同存在差异。因此,在目前研究中,我 们对每个肿瘤DNA标本采用相同的实验条件,以 达到不同标本之间相对甲基化率的有效可比性。
在一项RARβ基因功能性研究的实验中证实[9], RARβ基因具有与TP53基因一样抑制肿瘤生长的作 用。近年来,一些研究阐述了在乳腺恶性组织细胞 中RARβ基因mRNA的表达水平下降,而在正常乳 腺上皮细胞RARβ基因有表达,这些发现都表明了 RARβ基因mRNA表达的缺失可能是乳腺癌发生的 一个重要因素,并且是早期阶段[10]。RARβ基因位于 3p24上,该区域存在着乳腺癌杂合性丢失(loss of heterozygosity,LOH)的高概率(45%)[11]。但 是, LOH不能作为一个独立的因素抑制RARβ基因的表 达,因此,通过其进一步的研究证明,RARβ启动子 5’端区域的甲基化与乳腺癌发生具有重大的关系[12]。
本实验结果表明,乳腺癌组织及其癌旁组织 RARβ基因甲基化检出率差异有统计学意义,且乳腺癌中RARβ基因甲基化与肿瘤分期(P=0.010)、 肿瘤大小(P=0.012)呈正相关。这些发现把DNA 甲基化和乳腺癌的组织学特性关联起来,因而 显得重要。以往的研究结果亦有报道,基因CpG 岛甲基化与恶性程度高的乳腺癌相关,如ER、 E-cadherin、p16等基因[13, 14]。国外已有几个研究组 报道了CIMP+型大肠癌与恶性程度的相关性[15, 16, 17], 这表明这种DNA甲基化特异性表型的出现可能广 泛存在于人类癌症中。因此,我们认为RARβ基因 甲基化与恶性程度高的乳腺癌相关。
本研究还发现在浸润性非导管型乳腺癌中 RARβ基因甲基化率最高(50%),而Fackler等[18] 则认为RARβ基因甲基化对导管型乳腺癌和小叶型 乳腺癌同样重要。由于标本量小,本研究无法确 认RARβ基因甲基化与乳腺癌的某种组织分型是否 存在CIMP+表型。淋巴结转移、ER阴性、PR阴性 及HER2过度表达的癌组织中RARβ基因甲基化率 增高,但无统计学意义。所有这些发现均需要更 大的实验来研究,并且进一步明确乳腺癌的表型 特征是否与甲基化所致的基因失表达或其他机制 相关联。
鉴于RARβ基因甲基化与晚分期及直径>20 mm 乳腺癌的相关性,即RARβ基因甲基化与恶性程 度高的乳腺癌相关,可以推测此基因的甲基化提 示预后差。而且在淋巴结转移的肿瘤中,RARβ 基因甲基化的乳腺癌患者生存率显著下降(P= 0.040)。既往有研究报道CDH1和CCND2基因甲 基化提示乳腺癌患者预后差[19, 20],Widschwendter 等[21]亦发现ER基因甲基化具有相似的预后价值, 而且与内分泌治疗和化疗相关。因此与以往的研 究结果一致,我们认同基因甲基化对乳腺癌的预 后具有重要作用,虽然证据较薄弱,关于乳腺癌 DNA甲基化特异性表型的存在与否需要更多的病 例和其他基因加入研究来进一步明确。
总之,本研究证实,RARβ基因甲基化仅存在 于乳腺癌组织中,正常乳腺组织中无一例。乳腺 癌中RARβ基因甲基化与肿瘤分期和肿瘤大小的 相关性,表明RARβ基因甲基化在判定肿瘤预后起 重要作用。尤其是淋巴结转移的乳腺癌,RARβ基 因甲基化提示预后差。RARβ基因甲基化的研究对 于进一步明确乳腺癌的发病机制、早期诊断和预 后,以及乳腺癌DNA甲基化特异性表型的存在与 否具有重大的理论和临床意义。
| [1] | Anothaisintawee T, Teerawattananon Y, Wiratkapun C, et al. Risk prediction models of breast cancer: a systematic review of model performances[J]. Breast Cancer Res Treat,2012,133 (1):1-10. |
| [2] | Zhu W, Qin W, Hewett JE, et al. Quantitative evaluation of DNA hypermethylation in malignant and benign breast tissue and fluids[J]. Int J Cancer,2010, 126(2):474-82. |
| [3] | Cho KW, Kwon HJ, Shin JO,et al. Retinoic acid signaling and the initiation of mammary gland development[J]. Dev Biol,2012, 365 (1):259-66. |
| [4] | Salazar MD, Ratnam M, Patki M, et al. During hormone depletion or tamoxifen treatment of breast cancer cells the estrogen receptor apoprotein supports cell cycling through the retinoic acid receptor alpha1 apoprotein[J]. Breast Cancer Res,2011,13(1):R18. |
| [5] | Herman JG, Graff JR, My?h?nen S, et al. Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands[J]. Proc Natl Acad Sci U S A,1996,93 (18): 9821-6. |
| [6] | Paulin R, Grigg GW, Davey MW, et al. Urea improves efficiency of bisulphate-mediated sequencing of 5’-methylcytosine in genomic DNA[J]. Nucleic Acids Res,1998, 26 (21): 5009-10. |
| [7] | Zlobec I, Bihl M, Foerster A, et al. Comprehensive analysis of CpG island methylator phenotype (CIMP)-high, -low, and -negative colorectal cancers based on protein marker expression and molecular features[J]. J Pathol,2011, 225(3):336-43. |
| [8] | Soong R, Iacopetta BJ, Harvey JM, et al. Detection of p53 gene mutation by rapid PCR-SSCP and its association with poor survival in breast cancer[J]. Int J Cancer,1997, 74 (6): 642-7. |
| [9] | Lee X, Si SP, Tsou HC, et al. Cellular aging and transformation suppression: a role for retinoic acid receptor beta 2[J]. Exp Cell Res, 1995, 218 (1):296-304. |
| [10] | Sun J, Xu X, Liu J, et al. Epigenetic regulation of retinoic acid receptorβ2 gene in the initiation of breast cancer[J]. Med Oncol, 2011, 28(4):1311-8. |
| [11] | Lewis CM, Cler LR, Bu DW, et al. Promoter hypermethylation in benign breast epithelium in relation to predicted breast cancer risk[J]. Clin Cancer Res, 2005, 11(1):166-72. |
| [12] | Yang Q, Mori I, Shan L, et al. Biallelic inactivattion of retinoic acid receptor beta2 gene by epigenetic change in breast cancer[J]. Am J Pathol, 2001,158(1):299-303. |
| [13] | Yan PS, Perry MR, Laux DE, et al. CpG island arrays: an application toward deciphering epigenetic signatures of breast cancer[J]. Clin Cancer Res, 2000, 6(4): 1432-8. |
| [14] | Nass SJ, Herman JG, Gabrielson E, et al. Aberrant methylation of the estrogen receptor and E-cadherin 5’CpG islands increases with malignant progression in human breast cancer[J]. Cancer Res, 2000 ,60 (16): 4346-8. |
| [15] | Toyota M, Ahuja N, Ohe-Toyota M,et al. CpG island methylator phenotype in colorectal cancer[J]. Proc Natl Acad Sci U S A, 1999, 96 (15): 8681-6. |
| [16] | Hawkins N, Norrie M, Cheong K, et al. CpG island methylation in sporadic colorectal cancers and its relationship to microsatellite instability[J]. Gastroenterology, 2002, 122 (5): 1376-87. |
| [17] | van Rijnsoever M, Grieu F, Elsaleh H, et al. Characterisation of colorectal cancers showing hypermethylation at multiple CpG islands[J]. Gut, 2002, 51 (6):797-802. |
| [18] | Fackler MJ, McVeigh M, Evr on E, et al. DNA methylation of RASSF1A, HIN-1, RAR-beta, Cyclin D2 and Twist in in situ and invasive lobular breast carcinoma[J]. Int J Cancer, 2003, 107(6): 970-5. |
| [19] | Yang X, Yan L, Davidson NE. DNA methylation in breast cancer[J]. Endocr Relat Cancer, 2001, 8 (2): 115-27. |
| [20] | Mehrotra J, Ganpat MM, Kanaan Y,et al. Estrogen receptor/ progesterone receptor-negative breast cancers of young AfricanAmerican women have a higher frequency of methylation of multiple genes than those of Caucasian women[J]. Clin Cancer Res, 2004, 10 (6): 2052-7. |
| [21] | Widschwendter M, Siegmund KD, Müller HM, et al. Association of breast cancer DNA methylation profiles with hormone receptor status and response to tamoxifen[J]. Cancer Res,2004,64 (11):3807-13. |
 2014, Vol.41
2014, Vol.41




