原发性肝癌是临床上最常见的恶性肿瘤之 一,其发生发展过程涉及到癌基因、肿瘤抑制基 因、凋亡调节基因和DNA修复调节基因等多种基 因的异常。羧基末端结合蛋白(C-terminal binding protein 1,CtBP1)是一个癌基因,CtBP能作为转 录辅抑制因子抑制多个肿瘤抑制基因的转录,并 且与肿瘤的生物学行为密切相关[ 1 ]。我们前期研究 发现肝细胞肝癌组织中CtBP1表达上调;肝细胞肝 癌组织中其表达与肿瘤的分级相关[ 2 ]。为进一步阐 明CtBP1在肝细胞肝癌中的作用及机制,本研究通 过RNAi技术抑制HepG2细胞中CtBP1的表达,观 察CtBP1表达下调对细胞增殖、细胞周期与凋亡的 影响,为肝细胞肝癌发生发展的机制提供理论和 实验基础。 1 材料与方法 1.1 材料
高糖型DMEM培养液购自Hyclone公司, control siRNA-A、CtBP1 siRNA(h)、CtBP(C-1) 均购自Santa Cruz公司,X-tremeGENE siRNA Transfection Reagent购自Roche公司,Opti-MEM-I Medium购自Gibco公司,MTT购自Sigma公司, DMSO购自Amresco公司。 1.2 细胞株及其体外培养
人肝癌细胞株HepG2由桂林医学院科学实验 中心保存,用含10%胎牛血清及1%的青霉素和1% 链霉素的高糖型DMEM培养液培养于37℃、5% CO2、饱和湿度的培养箱中。 1.3 CtBP1 siRNA转染
对数生长期的HepG2细胞接种于6孔细胞培养 板或96孔培养板中,培养过夜至细胞融合度约70% 时转染,设空白对照组、转染试剂对照组、阴性 序列对照组和干扰组,每组设重复3孔。简述如 下:细胞1×105/ml接种于培养板中(六孔板2 ml, 96孔板100 μl);无抗生素DMEM培养液中培养过 夜至细胞融合度约70%时,转染细胞,siRNA的最 终浓度为20 pmol/ml(六孔板加入总体积100 μl的 siRNA-转染试剂混合物,96孔板15 μl);转染8 h 后换成完全培养液,培养至相应的时间用于后续 相关指标检测。 1.4 Western blot检测CtBP1的表达
转染后72 h收集细胞,加入含有PMSF及蛋白 酶抑制剂的RIPA裂解液,充分裂解后,4℃下12000 r/min离心30 min,取上清液即为蛋白样品。BCA 蛋白浓度测定法对蛋白样品进行定量。取30 μg 总蛋白进行SDS聚丙烯酰胺凝胶电泳,半干转膜 法转膜,室温条件下用牛奶封闭液封闭1h,一抗 CtBP(C-1)(用含5%脱脂奶粉的TBST配制)4℃ 孵育过夜,洗膜,二抗辣根酶标记山羊抗小鼠IgG (H+L)(用含5%脱脂奶粉的TBST配制)室温 孵育1 h,洗膜,发光、暴光、显影、定影,对X 光胶片扫描记录,测定各条带灰度值,蛋白相对 表达量=目的蛋白条带灰度值/内参照蛋白条带灰 度值。蛋白表达抑制率(%)=(1-干扰组相对 表达量/空白对照组相对表达量)×100%。 1.5 MTT法检测转染后HepG2细胞的增殖情况
细胞接种于96孔板中,分组及转染方法同1.3, 每组设6个复孔。分别于转染24、48、72、96 h 后,弃去培养液,PBS缓冲液冲洗2次,每孔加入 0.5%MTT溶液,置细胞培养箱中培养4 h后取出, 弃上清液,每孔加入150 μl DMSO,低速振荡10 min,待结晶物充分溶解后,酶标仪上于490 nm波 长处测量各孔的吸光度值(OD值),并以时间为 横坐标,OD值为纵坐标,绘制细胞生长曲线,按 生长抑制率=(1-实验组平均OD值/空白对照组平 均OD值)×100%计算转染后细胞生长抑制率。 1.6 流式细胞术检测HepG2细胞的细胞周期分布 及凋亡
细胞接种于6孔细胞培养板,分组及转染方法 同1.2,每组设3复孔,于转染后48 h收集细胞,用 4℃预冷的70%乙醇溶液重悬,4℃固定过夜,PI染 色法进行流式细胞术检测。以DNA含量检测细胞 周期分布和凋亡情况。 1.7 统计学方法
应用SPSS17.0统计软件。数据用均数±标准差 (x±s)表示,数据采用单因素方差分析,P<0.05 为差异具有统计学意义。 2 结果 2.1 CtBP1 siRNA转染抑制CtBP1蛋白的表达
与对照组相比,干扰组CtBP1的表达水平下 调,差异具有统计学意义(P<0.05),蛋白质抑 制率为57.80%,对照组之间的差异无统计学意义 (P>0.05),见图1。
 |
*:compared with control group,P<0.05
图1 转染后各组HepG2细胞CtBP1的表达
Figure 1 CtBP1 expression after transfected with siRNA in
HepG2 cells |
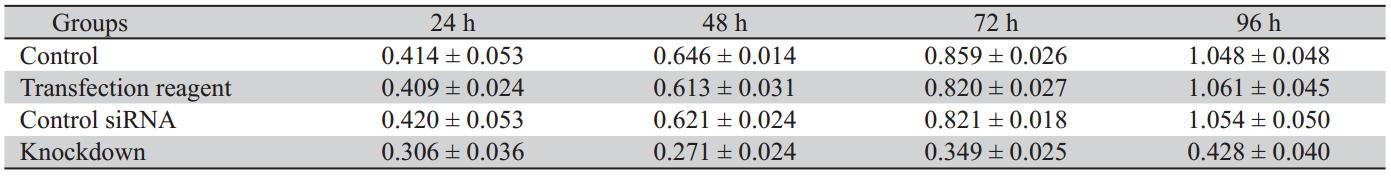
转染后24、48、72、96 h,干扰组OD值明 显低于对照组的OD值,差异具有统计学意义(<0.05)。干扰组细胞生长受到抑制,生长抑 制率在转染后24、48、72、96 h分别为22.34%、 52.15%、53.97%和54.77%,细胞生长曲线低平、 上升缓慢,对照组之间的差异不具有统计学意义 (P>0.05),见表1和图2。
|
|
表1 转染CtBP1 siRNA后各组HepG2细胞的OD值(x±s, n=5) Table 1 Optical density value of HepG2 cells detected by MTT after CtBP1 knockdown (x±s, n=5) |
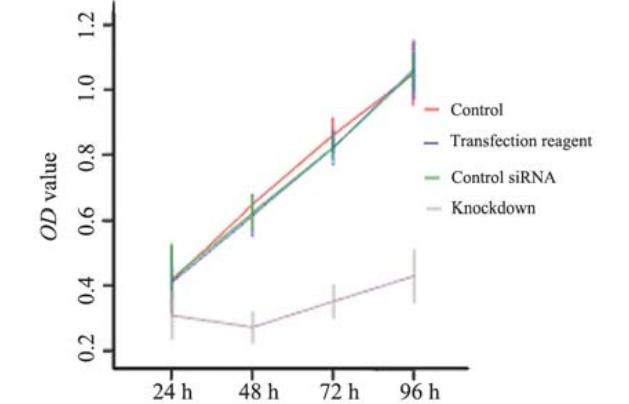 |
图2 转染CtBP1 siRNA后各组HepG2细胞的增殖情况 Figure 2 Proliferation curves of HepG2 cells after transfection with CtBP1 siRNA |
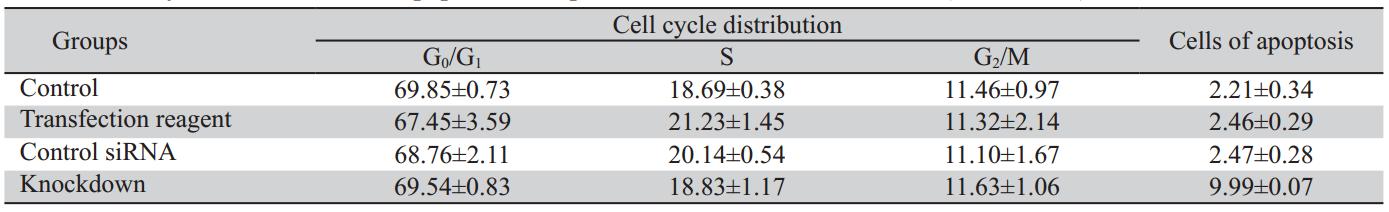
及凋亡 四个组之间细胞周期各时相细胞数的差异 无统计学意义(P>0.05),未观察到CtBP1 siRNA(h)对HepG2细胞的细胞周期分布有影响作 用,但观察到CtBP1 siRNA(h)对HepG2细胞有促凋 亡作用,与对照组相比,干扰组的细胞凋亡率显 著升高,差异具有统计学意义(P<0.05),对照 组之间的差异不具有统计学意义(P>0.05),见 图3、表2。
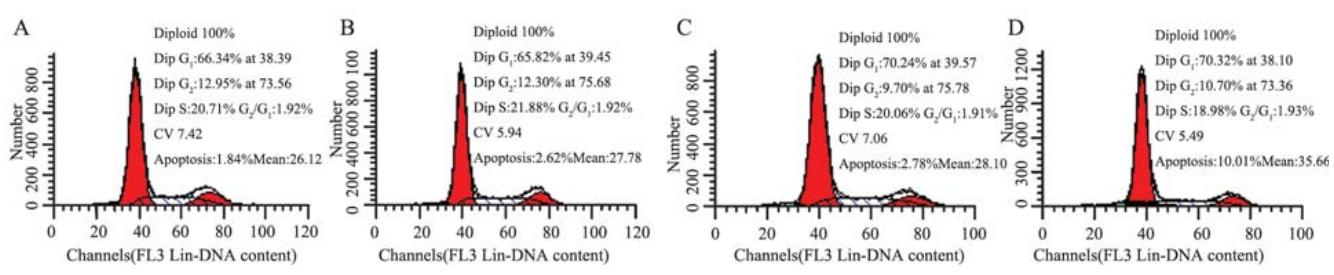 |
A:control group;B:transfection reagent group;C:control siRNA group;D:knockdown group
图3 转染CtBP1 siRNA后各组HepG2细胞的细胞周期变化及凋亡情况
Figure 3 Cell cycle distribution and apoptosis cells of HepG2 cells after transfection with CtBP1 siRNA |
|
|
表2 转染CtBP1 siRNA后各组HepG2细胞的细胞周期变化及凋亡情况(%,x±s, n=3) Table 2 Cell cycle distribution and apoptosis of HepG2 cells after CtBP1 knockdown(%,x±s, n=3 |
肝癌是常见的恶性肿瘤,死亡人数占到人类 恶性肿瘤的第三位[ 3 ]。其中肝细胞肝癌占到肝癌总 数的75%,肝细胞肝癌的病因包括肝炎病毒、酗 酒、代谢异常和环境毒物等[ 4 ]。发病机制涉及到多 个肿瘤相关基因的异常表达及其参与的多个信号 通路的异常[ 5 ]。
CtBP1是最近发现的一个癌基因[ 6 ]。CtBP1能 与腺病毒E1A蛋白结合,从而解除E1A蛋白羧基末 端的肿瘤抑制作用[ 7 ]。深入研究发现CtBP1可以作 为转录辅抑制因子抑制多个肿瘤抑制基因、上皮 细胞黏附分子的表达,从而调节多种细胞生物学 行为,包括上皮-间质转化(epithelial-mesenchymal transition,EMT)、细胞凋亡、细胞周期进程、 迁移、侵袭、血管生成及有丝分裂,从而促进肿 瘤的发生发展[ 8, 9 ]。
应用RNAi抑制CtBP1基因表达后,人乳腺细胞株MDA-MB-231及MCF-7的凋亡增加、增殖能力下 降、细胞有丝分裂减少[ 8 ];人 结 肠 癌 细 胞 株HCT116 的CtBP1表达下调可促进细胞凋亡[ 9 ]。我们前 期 研 究,发现肝细胞肝癌组织中CtBP1表达上调,肝细 胞肝癌组织中其表达与肿瘤的分级相关[ 2 ]。
为进一步明确CtBP1在肝细胞肝癌中的具体作 用机制,本研究在CtBP1高表达的肝细胞肝癌细 胞HepG2中,采用CtBP1 siRNA下调其表达,并观 察细胞生物学行为改变。结果显示,CtBP1 siRNA 有效抑制了CtBP1蛋白的表达,转染72 h后抑制率 达57.80%。通过MTT法检测细胞增殖,发现干扰 组细胞增殖受到明显抑制,证实了肝细胞肝癌中 CtBP1下调能抑制肿瘤细胞生长,与乳腺癌[ 8 ]和结 肠癌[ 9 ]中的结果相一致。表明CtBP1在肝细胞肝癌 中表达上调可能促进肿瘤的生长,与前期研究发 现的CtBP1高表达者肿瘤分级较高的结果相符[ 2 ]。
流式细胞术分析细胞周期发现,CtBP1沉默48 h后,各组细胞在细胞周期各时相的分布无明显差 别;而凋亡细胞比例显著增加。表明HepG2细胞中, CtBP1表达下调导致细胞生长抑制的主要机制是通 过促进细胞凋亡而非细胞周期阻滞来实现的。 CtBP1作为转录辅抑制因子能抑制多个促凋亡 蛋白的表达,包括pmp-22 相关p53效应因子(p53-effector related to pmp-22,PERP)、PTEN、p21、 Bax和Noxa等[ 10 ]。HepG2细胞中CtBP1干扰导致 CtBP1表达下调,解除了CtBP1对其促凋亡靶基因 的抑制,因而促进了细胞的凋亡,其具体机制仍 需进一步深入研究。
研究表明,CtBP1能抑制p16启动子的活性, 使p16 的表达下调[ 11 ]。p16是一个细胞周期抑制因 子,使细胞周期被阻滞于G1期。CtBP1能通过对 p16的抑制而促进细胞周期进展。而本研究表明, HepG2细胞中CtBP1沉默对细胞周期分布无明显影 响,可能与HepG2细胞内p16基因的CpG岛高度甲 基化[ 12 ]而表达缺失有关:CtBP1沉默能解除对p16启 动的抑制,但对其CpG岛高度甲基化并无影响;同 时表明CtBP1对细胞周期的影响依赖于p16。
上述数据表明,HepG2细胞中CtBP1沉默通过 促进细胞凋亡而抑制细胞生长。CtBP1是肝细胞肝 癌的潜在治疗靶点。
| [1] | Boyd JM,Subramanian T,Schaeper U,et al.A region in the C-terminus of adenovirus 2/5 E1a protein is required for association with a cellular phosphoprotein and important for the negative modulation of T24-ras mediated transformation, tumorigenesis and metastasis[J].EMBO J, 1993,12(2):469-78. |
| [2] | Zeng SE,Xiao SJ,Zhang XL,et al.Expression of transcriptional corepressor CtBP1 and it’s target gene E-cadherin in hepatocellular carcinoma[J].Lin Chuang Yu Shi Yan Bing Li Xue Za Zhi,2010,26(6):655-7.[曾思恩,肖胜军,张小玲,等.肝细胞肝癌 中转录辅抑制因子CtBP1与其靶基因E-cadherin的表达[J].临床 与实验病理学杂志, 2010,26(6):655-7.] |
| [3] | Forner A,Llovet JM,Bruix J.Hepatocellular carcinoma[J].Lancet, 2012,379(9822):1245-55. |
| [4] | Llovet JM, Burroughs A, Bruix J. Hepatocellular carcinoma[J]. Lancet, 2003, 362(9399): 1907-17. |
| [5] | Iakova P,Timchenko L,Timchenko NA.Intracellular signaling and hepatocellular carcinoma[J].Semin Cancer Biol,2011, 21(1):28-34. |
| [6] | Schaeper U,Boyd JM,Verma S,et al.Molecular cloning and characterization of a cellular phosphoprotein that interacts with a conserved C-terminal domain of adenovirus E1A involved in negative modulation of oncogenic transformation[J].Proc Natl Acad Sci U S A,1995,92(23):10467-71. |
| [7] | Schaeper U, Subramanian T, Lim L, et al. Interaction between a cellular protein that binds to the C-terminal region of adenovirus E1A (CtBP) and a novel cellular protein is disrupted by E1A through a conserved PLDLS motif[J]. J Biol Chem, 1998, 273(15):8549-52. |
| [8] | Bergman LM,Birts CN,Darley M,et al.CtBPs promote cell survival through the maintenance of mitotic fidelity[J].Mol Cell Biol, 2009,29(16):4539-51. |
| [9] | Bergman LM,Blaydes JP.C-terminal binding proteins: emerging roles in cell survival and tumorigenesis[J].Apoptosis,2006, 11(6):879-88. |
| [10] | Chinnadurai G.The transcriptional corepressor CtBP: a foe of multiple tumor suppressors[J].Cancer Res,2009, 69(3):731-4. |
| [11] | Mroz EA,Baird AH,Michaud WA,et al.COOH-terminal binding protein regulates expression of the p16INK4A tumor suppressor and senescence in primary human cells[J].Cancer Res,2008, 68(15):6049-53. |
| [12] | Zang JJ,Xie F,Xu JF,et al.P16 gene hypermethylation and hepatocellular carcinoma: a systematic review and metaanalysis[J].World J Gastroenterol, 2011, 17(25):3043-8. |
 2014, Vol.41
2014, Vol.41