Bioinformatic and Mendelian Randomization Analyses of Correlation Between Differentially Expressed Genes and Prognosis of Oral Squamous Cell Carcinoma Patients
-
摘要:目的
通过生物信息学和孟德尔随机化分析口腔鳞状细胞癌(OSCC)中的差异表达基因,探讨其与OSCC预后的相关性。
方法TCGA数据库和数据集GSE138206筛选OSCC共同的差异基因,并利用孟德尔随机化分析它们之间的关系,进一步通过Cox风险回归分析差异基因的预后价值,对预后价值高的基因进行单基因差异分析后进一步评估其生物学功能。
结果两个数据库共筛选出147个共同差异基因,双样本孟德尔随机化结果显示GREM2与口腔鳞状细胞癌风险增加有关,SH3BGRL2与其风险减小有关,此外DKK1、CCL11和HOXC6是OSCC预后的独立标志基因,而DKK1的预测结果与实际结果的一致性最好。KEGG富集通路显示DKK1可能参与花生四烯酸代谢与亚油酸代谢。此外,DKK1与Tgd和Th2细胞呈正相关,与PDC、Cytotoxic、Mast、CD8 T、TFH、B、T和Th17细胞呈负相关。
结论GREM2与OSCC风险增加有关、DKK1在OSCC中高表达且与不良预后相关,其可能参与调控OSCC中花生四烯酸与亚油酸代谢及免疫细胞浸润进程,为后续研究奠定了基础。
Abstract:ObjectiveDifferentially expressed genes in oral squamous cell carcinoma (OSCC) were subjected to bioinformatic and Mendelian randomization analyses to elucidate their prognostic significance in OSCC.
MethodsThe TCGA database and dataset GSE138206 were used to screen the common differential genes of OSCC, and their relationship was analyzed by using Mendelian randomization. The prognostic value of differential genes was further analyzed by Cox risk regression. The biological function of genes with high prognostic value was further evaluated by single gene differential analysis.
ResultsA total of 147 common differential genes were screened from the two databases. Results of two-sample Mendelian randomization showed that GREM2 was associated with the increased risk of OSCC. In addition, SH3BGRL2 was associated with a decreased risk of OSCC, and DKK1, CCL11, and HOXC6 were considered as independent prognostic markers of OSCC. The predicted results of DKK1 were consistent with the actual results. KEGG enrichment analysis indicated the potential involvement of DKK1 in arachidonic acid and linoleic acid metabolism. Furthermore, DKK1 showed positive correlations with Tgd and Th2 cells, while displaying negative associations with PDC, Cytotoxic cells, Mast cells, CD8 T cells, TFH cells, B cells, T cells, and Th17 cells.
ConclusionGREM2 is associated with an increased risk of OSCC. DKK1 is highly expressed in OSCC and associated with poor prognosis, which may be involved in regulating the metabolism of arachidonic acid and linoleic acid and immune cell invasion in OSCC.
-
0 引言
口腔鳞状细胞癌(Oral squamous cell carcinoma, OSCC)是头颈部最常见的恶性肿瘤之一,具有较强侵袭性和异质性。根据全球癌症观察站(GCO)的预测,到2040年,OSCC的发病率将增加约40%,死亡率也会相应上升[1]。尽管早期防治可行,但OSCC的病理机制仍不明确且早期症状隐匿,在大多数患者中只有在进展时才能确诊,并已处于晚期。虽然已提出各种治疗干预措施如化疗、放射治疗、免疫治疗和纳米医学来预防或治愈OSCC,但晚期OSCC患者5年总生存率仍然很低[2]。这主要归因于OSCC发生机制复杂且不确定,并缺乏可靠的诊断和治疗靶点[3]。
孟德尔随机化(Mendelian Randomization, MR)是一种利用遗传变异作为工具变量,来评估因果关系的统计方法[4]。其分析可以提供差异基因与OSCC之间因果关系的证据。传统的观察性研究往往难以区分因果关系和相关性,而MR分析通过使用遗传变异作为工具变量,有助于确定基因变异是否是OSCC的因果因素。最近一项研究中,使用孟德尔随机化法揭示了血液代谢物与OSCC之间的因果关系[5]。根据另一项孟德尔随机化研究,多种血清生物标志物可能与OSCC和口咽癌(OC/OPC)的风险相关[6]。因此,本研究采用孟德尔随机化法探讨OSCC差异基因与OSCC之间的关系。深入探讨OSCC患者的组织特征及其与预后相关因素,并以此为基础展开针对该癌种的有效治疗显得至关重要[7]。本研究利用癌症基因组图谱(TCGA)和高通量基因表达数据库(GEO)的差异基因,对其进行孟德尔相关分析以及评估预后,并分析该预测模型与OSCC患者临床病理特征之间的关联。同时,我们还探讨了这些预测性基因与肿瘤免疫微环境特征之间的联系,旨在为OSCC患者的临床治疗和预后改善提供有益指导。
1 资料与方法
1.1 数据来源
口腔鳞状细胞癌的GWAS数据来自于IEU Open GWAS数据库(https://gwas.mrcieu.ac.uk/datasets/ebi-a-GCST012239/)。通过该数据库下载了来源于口腔鳞状细胞癌的GWAS汇总数据,其ID为ebi-a-GCST012239,涉及6 758 174个SNPs。
1.2 筛选差异基因
通过GEOquery包从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中下载GSE138206基因表达微阵列数据集。该芯片基于GPL570,研究选取6位口腔鳞状细胞癌患者和6位健康个体进行差异基因分析(Differentially expressed genes, DEGs)。从TCGA数据库(https://portal.gdc.cancer.gov)下载并整理TCGA-HNSC(头颈部鳞状细胞癌)数据以及临床数据,在临床信息中保留属于口腔鳞状细胞癌部位(Alveolar Ridge, Base of tongue, Buccal Mucosa, Floor of mouth, Hard Palate, Oral Cavity, Oral Tongue)样本,剔除非口腔鳞状细胞癌部位(Hypopharynx, Larynx, Lip, Oropharynx, Tonsil)样本。差异基因的纳入标准为|LogFC|>2和P.adj<0.05,将不同数据库获得的差异基因进行交集。
1.3 工具变量筛选
在MR分析中,为了确保足够数量的SNP,设定阈值为P<1×10−6进行筛选。随后,去除具有强连锁不平衡(LD)的IVs(r2<0.001, kb=10000)。接着,计算F统计量,公式为F=[R2/(1−R2)]×(N-K-1)/K,R2=2×MAF×(1−MAF)×(β/sd)2,其中N表示与暴露因子相关的样本量,K表示工具变量的数量,R2表示可以由工具变量解释的暴露因子变化的比例。若F<10考虑为弱工具变量,应剔除。
1.4 双样本MR分析方法
为了探究差异基因与口腔鳞状细胞癌的因果关系,在本研究中采取双样本MR分析方法。工具变量SNP应满足“与暴露因素相关”的假设(①工具变量与暴露因素相关;②工具变量不会受到混杂因素的影响;③工具变量只能通过暴露因素影响结局因素)。使用“TwoSampleMR”进行MR分析,应用各种MR分析方法来校验差异基因与OSCC之间因果关系的准确性,包括逆方差加权法(Inverse-variance weighted, IVW)、MR-Egger回归和加权众数法(Weighted mode, WM),其中IVW方法占主导地位,因为其具有更高的统计功效。IVW方法的P<0.05被认为提示潜在的因果关系。采用Egger-intercept检验评估水平多效性的存在,通过留一法进行敏感性分析,即剔除单个离群SNP对MR结果不造成明显影响,用散点图、森林图和漏斗图来展示结果。
1.5 Cox风险回归分析
使用survival包进行比例风险假设检验并进行拟合生存回归,用于比较交集基因高低组之间的预后情况,结果用survminer包以及ggplot2包进行可视化。采用单因素和多因素Cox回归分析确定患者OS的独立预测因素,并基于独立预后分析构建列线图,研究的变量包括临床与病理分期、T分期、N分期、性别、年龄、吸烟以及预后基因的表达等。
1.6 单基因差异分析
在OSCC样本中,将上述分析出的基因表达值分成高低表达组,分析两组中的差异分子。以|LogFC|>1和P.adj<0.05为条件筛选,以筛选该分子可能关联的分子。进一步对有关联的分子进行KEGG富集分析,利用提供的分子log2FC值通过GOplot包计算每个富集条目对应的zscore值。
1.7 免疫浸润分析
根据TCGA-OSCC数据中关键分子在所有样本中的中位数,将样本分为高低表达组。基于R包-GSVA[1.46.0][8]中提供的ssGSEA算法,计算每个样本的免疫细胞分数,并进行高低分组间的比较分析,分析其是否有统计学差异。利用Spearman方法对上述有统计学意义的免疫细胞与关键基因进行相关性分析[9]。
1.8 统计学方法
当两组需要满足正态性和方差齐性时采用T test, 亦称student t检验;当两组仅满足正态而不满足方差齐性的要求时,可以选择用Weltch`t test方法进行两组的比较;当两组不满足正态性的情况时采用秩和检验,该方法是一个非参的假设检验方法。
2 结果
2.1 差异表达基因的筛选结果
在TCGA-OSCC数据库中,采用|LogFC|>2和P.adj<0.05的标准,我们鉴定出1 826个差异基因。其中,
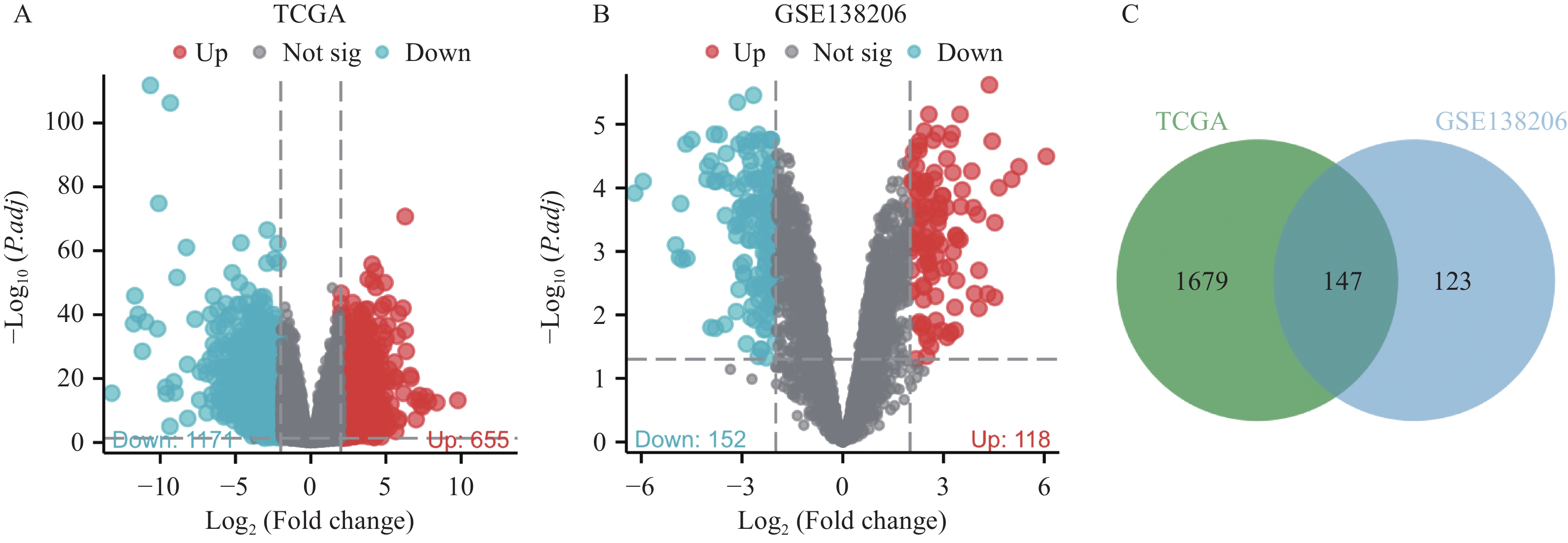
1171 个基因表达下调,655个基因表达上调(图1A)。在GSE138206数据集中,我们通过设定|LogFC|>2和P.adj<0.05的条件,筛选出270个差异基因。在270个差异基因中,其中118个上调,152个下调(图1B)。两个数据库对比发现,共有147个基因在两者中均表现为差异表达(图1C)。2.2 差异基因与OSCC的关系
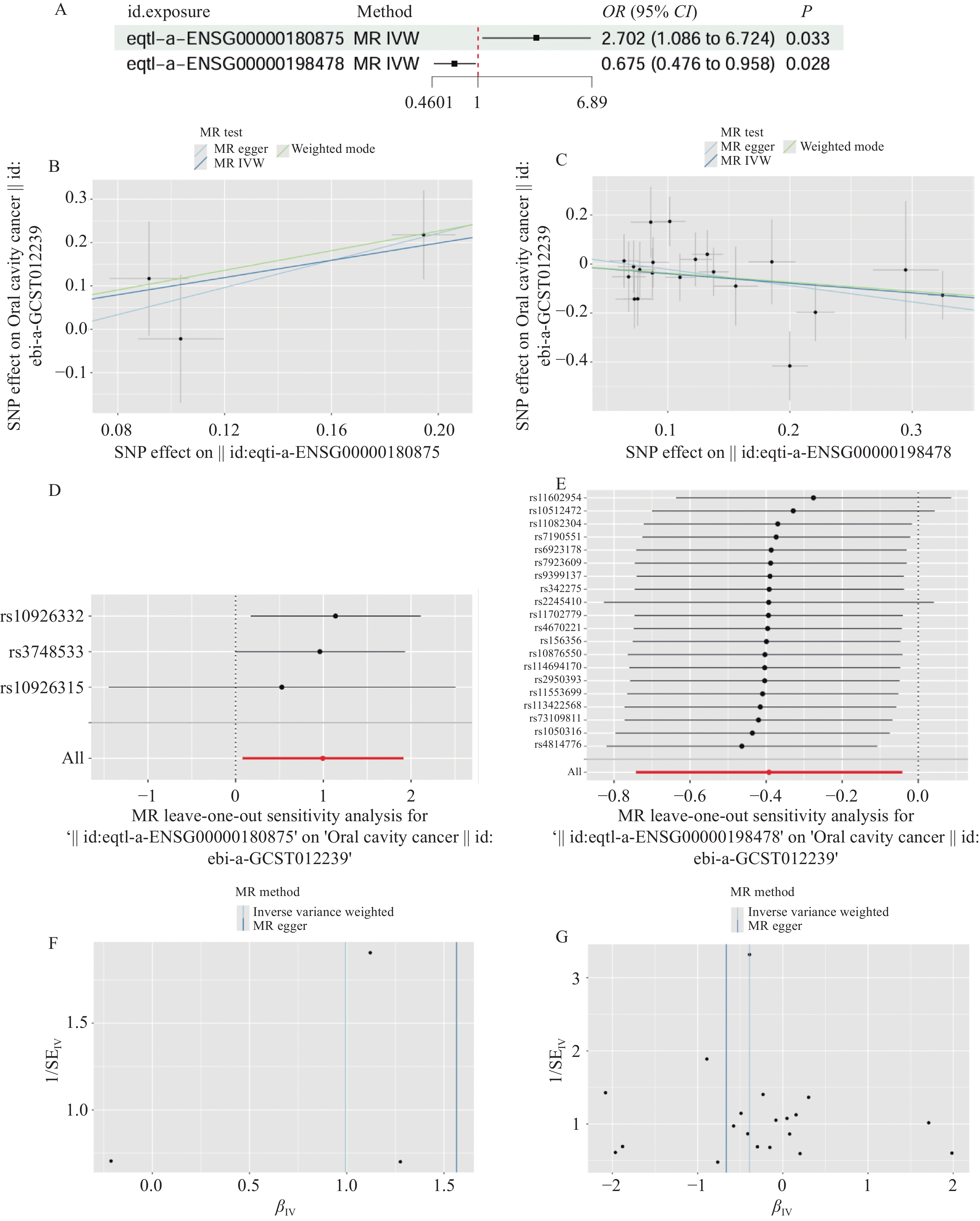
MR分析的IVW模型结果如图2A显示,差异基因GREM2 (eqtl-a-ENSG00000180875;OR=2.702;95%CI: 1.086~6.724;P=0.033)与OSCC风险增加有关,SH3BGRL2(eqtl-a-ENSG00000198478;OR=0.675;95%CI: 0.476~0.958;P=0.028)与OSCC风险减小有关(图2B)。逆方差加权法(IVW)、MR-Egger回归和加权众数法方法表明,差异基因的SNP与OSCC关系显著。其中,IVW方法是主要分析方法。对主要的IVW分析法和MR-Egger回归法采取异质性检验,检验结果中P>0.05则认为纳入的工具变量不存在异质性,可以忽略异质性对因果效应估计产生的影响,Egger-intercept进行水平多效性检验,P值均大于0.05,则表明其均不存在水平多效性。结果显示P值均大于0.05(表1),这证明了结果均不存在异质性和水平多效性,表明了因果关系的稳健,结果是可信的。留一法提示结果稳定,剔除单个离群SNP对MR结果不造成明显影响(图2D、E)。漏斗图左右两侧对称,提示结果稳定(图2F、G)。
![]() 图 2 GREM2和SH3BGRL2与OSCC因果关系分析结果Figure 2 Causal relationship between GREM2, SH3BGRL2 and OSCCA: the IVW results of the relationship between GREM2, SH3BGRL2 and OSCC; B: the scatter plot of GREM2 and OSCC; C: the scatter plot of SH3BGRL2 and OSCC; D: the leave one out of the relationship between GREM2 and OSCC; E: the leave one out of the relationship between SH3BGRL2 and OSCC; F: the funnel plot of the relationship between GREM2 and OSCC; G: funnel plot showing the relationship between SH3BGRL2 and OSCC.表 1 差异基因与OSCC的关系的异质性和水平多效性检验Table 1 Heterogeneity and horizontal pleiotropy tests of relationship between differential genes and OSCC
图 2 GREM2和SH3BGRL2与OSCC因果关系分析结果Figure 2 Causal relationship between GREM2, SH3BGRL2 and OSCCA: the IVW results of the relationship between GREM2, SH3BGRL2 and OSCC; B: the scatter plot of GREM2 and OSCC; C: the scatter plot of SH3BGRL2 and OSCC; D: the leave one out of the relationship between GREM2 and OSCC; E: the leave one out of the relationship between SH3BGRL2 and OSCC; F: the funnel plot of the relationship between GREM2 and OSCC; G: funnel plot showing the relationship between SH3BGRL2 and OSCC.表 1 差异基因与OSCC的关系的异质性和水平多效性检验Table 1 Heterogeneity and horizontal pleiotropy tests of relationship between differential genes and OSCCid.exposure id.outcome Heterogeneity Horizontal pleiotropy P IVW Q P MR-Egger Q P Egger-intercept eqtl-a-ENSG00000180875 ebi-a-GCST012239 0.66 0.42 0.75 eqtl-a-ENSG00000198478 0.56 0.55 0.41 2.3 Cox分险回归分析
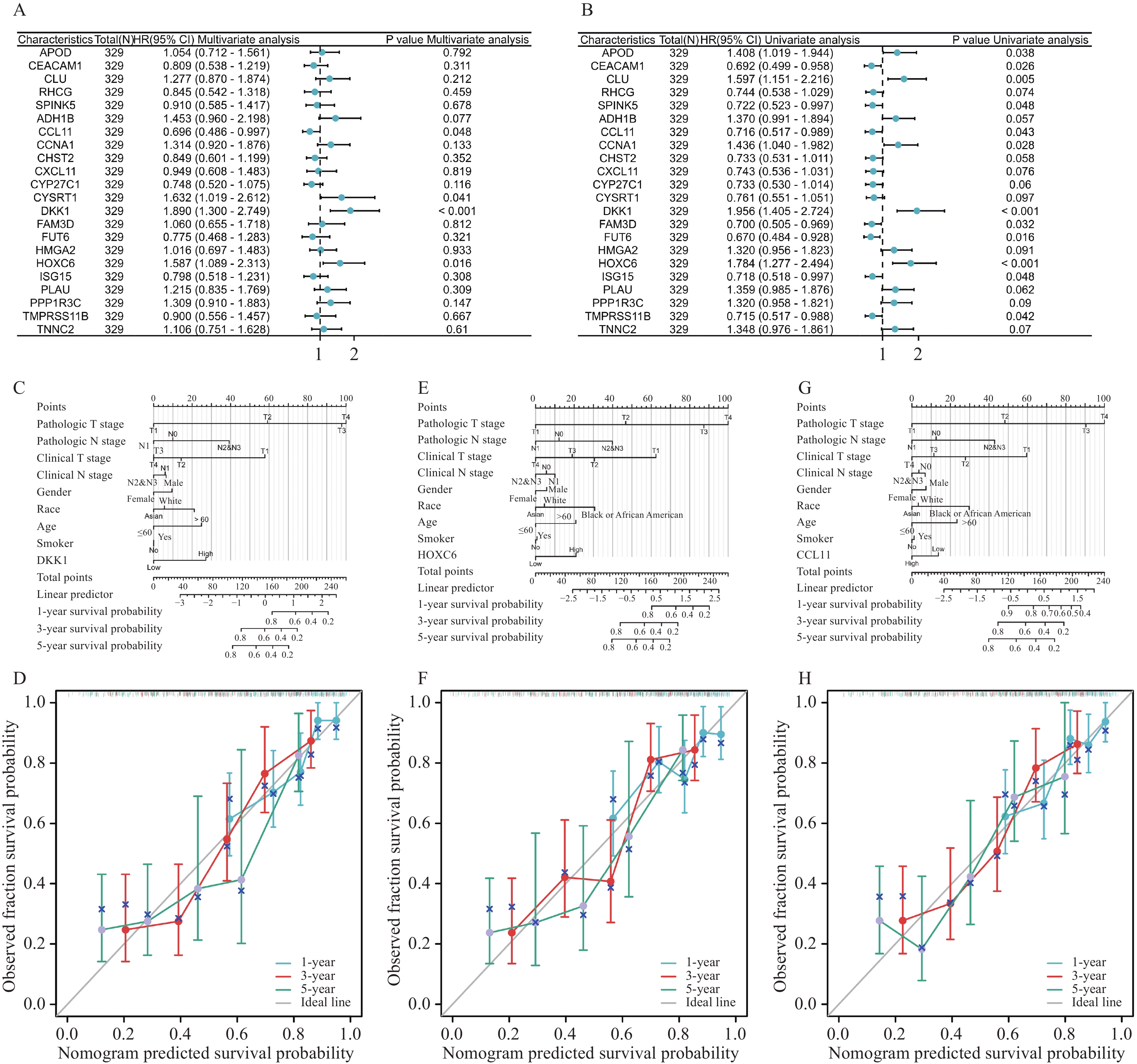
对上述的147个基因进行单因素分析,其P<0.1有22个基因。进一步进行多因素分析发现CCL11、HOXC6和DKK1同时满足单因素和多因素结果(图3A、B)。因此,CCL11、HOXC6和DKK1为OSCC患者预后的独立标志基因。随后,通过构建患者临床病理特征、性别、年龄、种族、吸烟以及CCL11、HOXC6和DKK1表达水平为参数的列线图模型。列线图模型的C指数分别为0.689、0.688、0.713(图3C-H)。其中DKK1的预测结果与实际结果的一致性最好。
2.4 预后分析
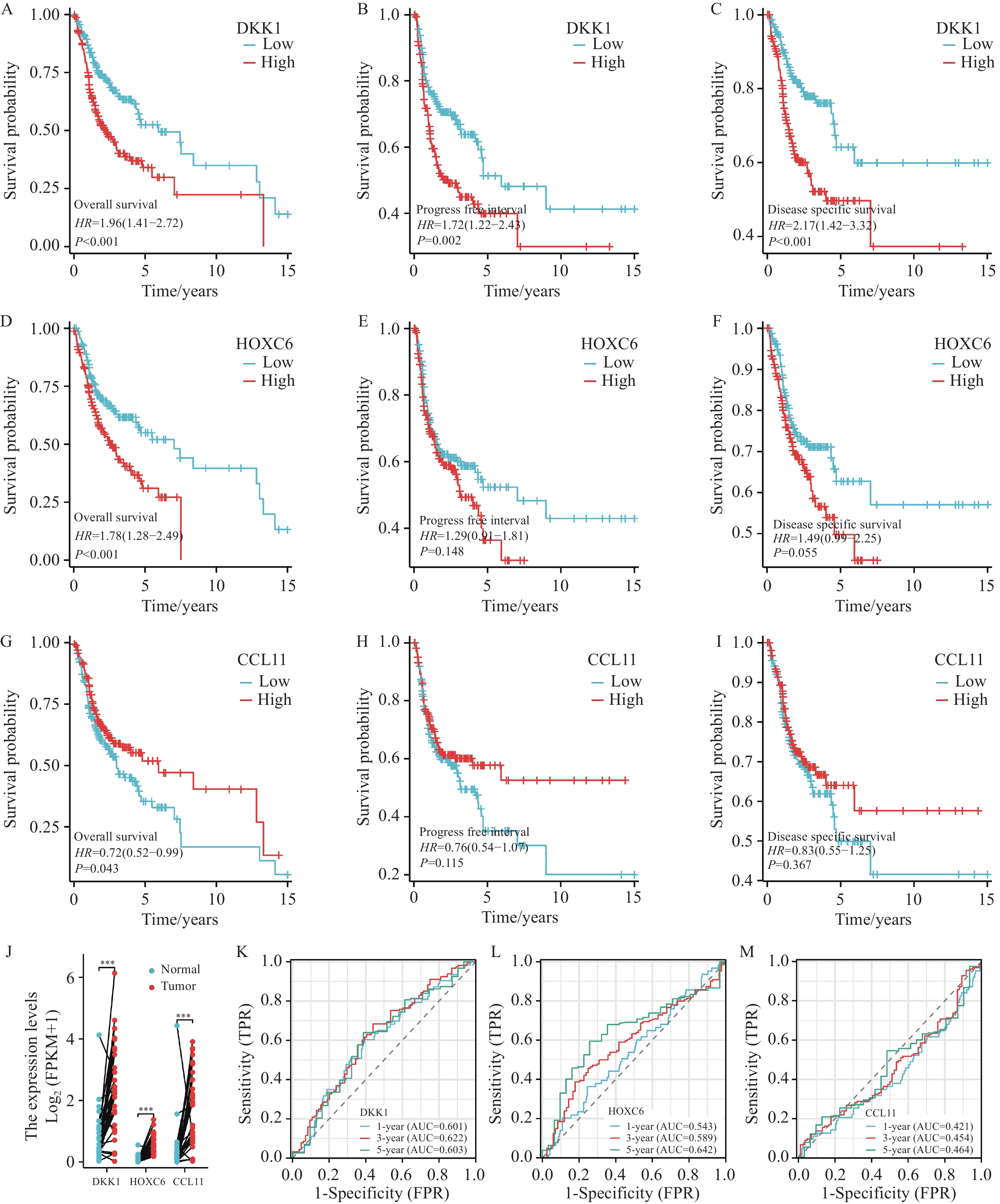
在OSCC中,DKK1、HOXC6和CCL11高表达。DKK1高表达与总体生存期(Overall survival, OS)、无进展间隔期(Progression free interval, PFI)、疾病特异性生存期(Disease specific survival, DSS)的不良预后有关,且差异有统计学意义,而HOXC6和CCL11只与OS不良预后有关。绘制时间依赖的ROC曲线,结果表明DKK1在OS期的1、3、5年有较好的预后性能;相较于DKK1的预后性能,HOXC6的预后性较差,CCL11的预后性能更差,见图4。
2.5 DKK1单基因差异分析
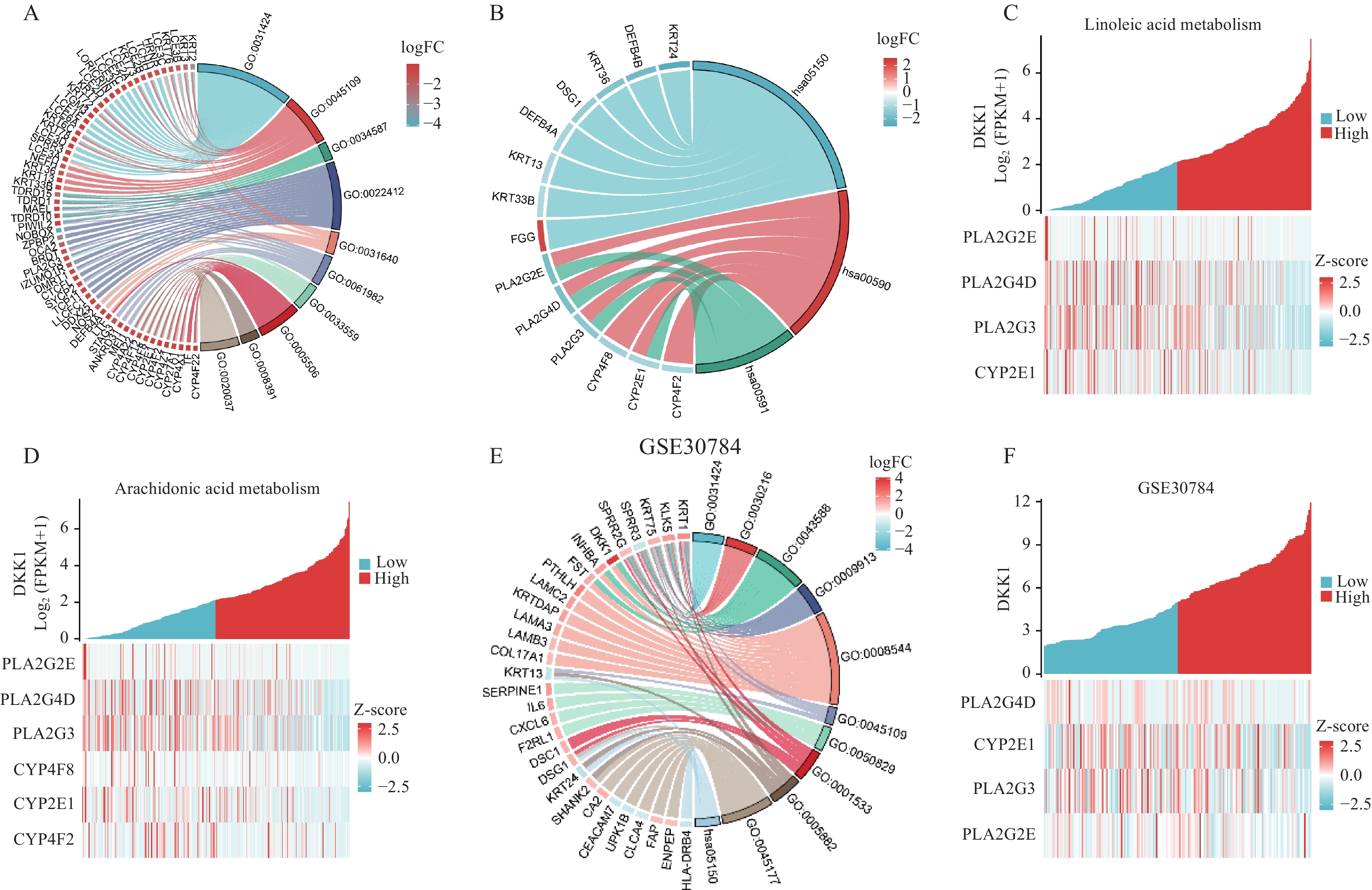
根据DKK1 mRNA表达的中位数将TCGA数据库中OSCC样本分为高表达组和低表达组,最终发现有258个蛋白编码类的基因显著下调,5个蛋白编码类的基因显著上调。进一步评估DKK1相关DEG的生物学功能,富集到金黄色葡萄球菌感染、花生四烯酸代谢、亚油酸代谢。参与的生物学过程包括角化作用、中间细丝组织、piRNA代谢过程、多细胞生物中涉及生殖的细胞过程、杀死另一生物体的细胞、减数分裂Ⅰ细胞周期过程、不饱和脂肪酸代谢过程等。此外,还发现上述富集到的条目可能对OSCC起到负调节作用(图5A、B)。其中,DKK1高表达组与花生四烯酸代谢和亚油酸代谢相关基因都呈负相关(图5C、D)。为了进一步验证上述发现,我们采用相同的方法对GSE30784数据集进行分析。分析结果再次证实,涉及的生物学过程包括表皮发育、角化作用、皮肤生长以及对革兰氏阴性菌的防御反应等。同样地,这些过程也参与了对抗金黄色葡萄球菌感染的途径(图5E)。此外,我们还观察到DKK1高表达组与亚油酸代谢相关基因之间存在显著的负相关性(图5F)。
![]() 图 5 生物学功能富集分析Figure 5 Biological function enrichment analysisA: biological process analysis; B: pathway enrichment analysis; C: co-expression heat map of genes related to DKK1 and linoleic acid metabolism; D: heat map of the co-expression of genes related to DKK1 and arachidonic acid metabolism; E: enrichment analysis of the GSE30784 dataset; F: co-expression heat map of genes related to linoleic acid metabolism in the GSE30784 dataset.
图 5 生物学功能富集分析Figure 5 Biological function enrichment analysisA: biological process analysis; B: pathway enrichment analysis; C: co-expression heat map of genes related to DKK1 and linoleic acid metabolism; D: heat map of the co-expression of genes related to DKK1 and arachidonic acid metabolism; E: enrichment analysis of the GSE30784 dataset; F: co-expression heat map of genes related to linoleic acid metabolism in the GSE30784 dataset.2.6 DKK1与免疫浸润的相关性分析
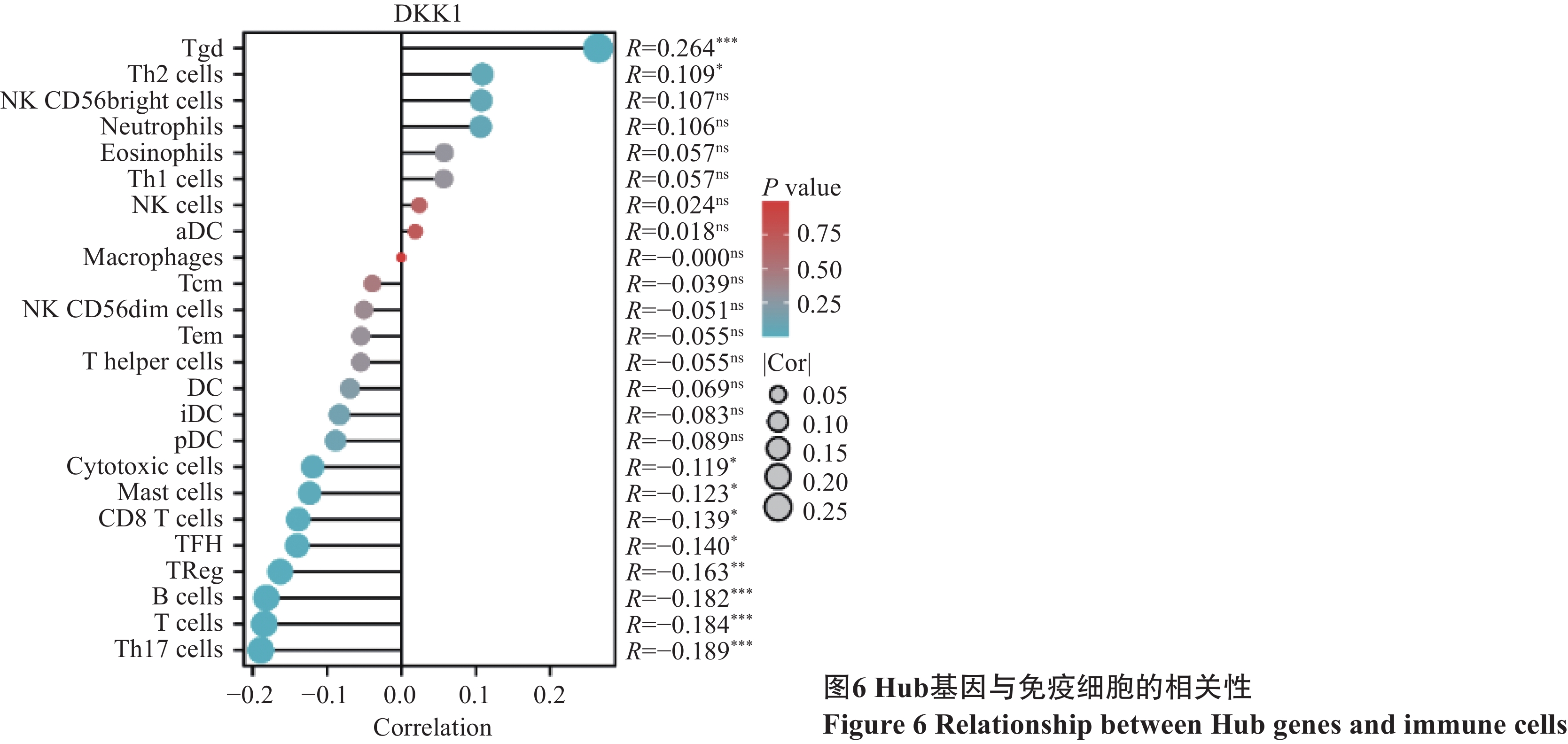
基于TCGA-OSCC数据中DKK1的中位数,将样本分为高、低表达组,然后分析DKK1与24种免疫细胞间的关系,最后发现DKK1与Tgd和Th2细胞呈正相关;DKK1与浆细胞样树突状细胞、细胞毒性细胞、肥大细胞、CD8 T细胞、Tfh滤泡辅助性T细胞、B细胞、T细胞和Th17细胞呈负相关(图6)。
3 讨论
OSCC是一种恶性肿瘤,不健康的生活方式如吸烟和饮酒会加剧OSCC的发展,并引起口腔疼痛、喉咙不适等症状,进而影响患者的食欲和营养摄入[10]。目前,OSCC治疗通常采用多学科综合治理方法,包括手术、化学治疗和放射治疗等,然而,患者的预后仍然很差[11]。异常基因表达可参与肿瘤发生,并与患者的预后有关[12]。本项研究旨在确定与OSCC预后相关的基因,并探索这些基因对生物过程和功能的潜在贡献。通过分析TCGA数据库中的OSCC数据以及GEO数据库中GSE138206数据集中差异表达基因,我们发现DKK1、CCL11、HOXC6是OSCC患者预后的独立标志基因,其中CCL1和HOXC6高表达只与OS的不良预后有关,而DKK1高表达与OS、DSS、PFI的不良预后都有关。因此,我们重点关注了DKK1在OSCC中的作用。Dickkopf相关蛋白1(Dickkopf-related protein 1, DKK1)是一种分泌型糖蛋白,作为Wnt/β-catenin信号通路的典型抑制剂,主要通过与LRP5/6受体竞争性结合来拮抗Wnt/β-catenin信号通路活性,并在细胞增殖、分化、癌变以及细胞凋亡、瘤细胞侵袭和转移等方面发挥调节作用[13]。在对DKK1单基因进行差异表达分析后,我们揭示DKK1在角化过程、中间丝组织架构、花生四烯酸代谢途径以及亚油酸代谢路径中发挥着关键作用。为了确定这一研究成果,我们采用了一致的分析手段,在GSE30784数据集中对DKK1基因进行了差异表达分析,并成功验证了我们的初步发现。HOXC6属于同源盒家族中高度保守的转录因子家族,在多细胞生物形态发生中扮演重要角色。越来越多的研究表明,DKK1和HOXC6在肿瘤发生和发展过程中具有关键功能[14]。它们在鳞状细胞癌如头颈鳞癌[15]和喉鳞状细胞癌[16]中高度表达,并被认为是致癌基因之一。值得注意的是,Qi等指出HOXC6可以通过DKK1/Wnt/β-catenin轴促进右半结肠癌(RCC)转移机制,这对于RCC治疗中分子靶点的探索提供了新思路[17]。这些数据标明,DKK1在肿瘤发生和发展中起关键作用。
肿瘤微环境中的免疫细胞在肿瘤的发生和发展中发挥着重要作用,并且与预后显著相关[18]。肥大细胞在免疫反应中扮演着重要角色。例如,在肿瘤微环境中,它们的活化状态和局部微环境决定了其促进或抑制肿瘤生长的具体作用。我们观察到高DKK1组OSCC中的肥大细胞显著减少,提示DKK1可能通过影响肥大细胞来调控OSCC的生长和进展。此外,在OSCC组织中,辅助性T细胞2和Tgd细胞在高DKK1组中增加,并与DKK1表达呈正相关,暗示DKK1可能通过促进这些特定免疫细胞群的分化和功能来推动特定的免疫反应。这些发现与Liu等的发现一致[19]。相比之下,DKK1与浆细胞样树突状细胞、细胞毒性细胞、肥大细胞、CD8 T细胞、滤泡辅助性T细胞、B细胞、T细胞和Th17细胞呈负相关,提示DKK1可能通过削弱肿瘤免疫监视和反应来促进肿瘤的生长和进展。上述结果揭示了DKK1在调节OSCC肿瘤免疫微环境中的关键作用,但是更深层次机制仍需进一步探究。
有研究表明与牙齿发育不全相关的基因变异可以作为癌症的预后或治疗靶点[20]。孟德尔结果显示GREM2与OSCC风险增加有关,研究表明GREM2是一种骨形态发生蛋白(BMP)拮抗剂,可能通过抑制BMP信号通路促进上皮间质转化,从而促进OSCC细胞的侵袭和转移。另一发现SH3BGRL2与OSCC风险减小有关。研究表明SH3BGRL2的表达在体外显著抑制胶质母细胞瘤细胞的生长、迁移和胶质瘤干细胞的自我更新以及体内肿瘤的生长[21],而且它被认为是透明细胞肾细胞癌中一种新的下调肿瘤抑制因子[22]。孟德尔随机化通过使用遗传变异作为工具变量,可以在一定程度上推断因果关系,而传统的生物信息学分析通常只能揭示相关性,不能直接证明因果关系。然而,值得注意的是,孟德尔随机化方法也存在一定的局限性,比如它依赖于足够数量的遗传变异作为工具变量,并且可能受到基因多效性的影响(即一个基因可能影响多个性状)。因此,孟德尔随机化与生物信息学分析通常是相互补充的两种研究手段。
综上所述,本研究运用生物信息学方法以及孟德尔随机化研究探索OSCC预后相关基因,并报道了关键基因DKK1可能与不良预后相关。具体而言,DKK1可能通过削弱肿瘤免疫监视和反应来促进肿瘤的增长和发展。生物信息学与孟德尔随机化结合能够发现新的治疗靶点,两者相互补充。然而,需要注意的是本研究未得到基础实验和临床试验的验证存在一定的局限性,因此,下一步计划在体外进行验证上述结果。
Competing interests: The authors declare that they have no competing interests.利益冲突声明:所有作者均声明不存在利益冲突。作者贡献:刘茂林:思路设计、数据收集及分析、文章撰写宋晓娜、刘雅琪、史舒瑄:数据收集及分析宋国华:思路设计、文章修改 -
表 1 差异基因与OSCC的关系的异质性和水平多效性检验
Table 1 Heterogeneity and horizontal pleiotropy tests of relationship between differential genes and OSCC
id.exposure id.outcome Heterogeneity Horizontal pleiotropy P IVW Q P MR-Egger Q P Egger-intercept eqtl-a-ENSG00000180875 ebi-a-GCST012239 0.66 0.42 0.75 eqtl-a-ENSG00000198478 0.56 0.55 0.41 -
[1] Tan Y, Wang Z, Xu M, et al. Oral squamous cell carcinomas: state of the field and emerging directions[J]. Int J Oral Sci, 2023, 15(1): 44. doi: 10.1038/s41368-023-00249-w
[2] Ng JH, Iyer NG, Tan MH, et al. Changing epidemiology of oral squamous cell carcinoma of the tongue: A global study[J]. Head Neck, 2017, 39(2): 297-304. doi: 10.1002/hed.24589
[3] Pagoni P, Dimou NL, Murphy N, et al. Using Mendelian randomisation to assess causality in observational studies[J]. Evid Based Ment Health, 2019, 22(2): 67-71. doi: 10.1136/ebmental-2019-300085
[4] Hu Z, Xu Z, Yue Q, et al. The role of blood metabolites in oral cancer: insights from a Mendelian randomization approach[J]. Front Oncol, 2024, 14: 1305684. doi: 10.3389/fonc.2024.1305684
[5] Liu W, Liu Y, Li P, et al. Causal association of serum biomarkers with oral cavity and oropharyngeal cancer: a mendelian randomization study[J]. BMC Oral Health, 2023, 23(1): 987. doi: 10.1186/s12903-023-03729-x
[6] Zhou W, Feng Y, Lin C, et al. Yin Yang 1-Induced Long Noncoding RNA DUXAP9 Drives the Progression of Oral Squamous Cell Carcinoma by Blocking CDK1-Mediated EZH2 Degradation[J]. Adv Sci (Weinh), 2023, 10(25): e2207549. doi: 10.1002/advs.202207549
[7] Khurshid Z, Zafar MS, Khan RS, et al. Role of Salivary Biomarkers in Oral Cancer Detection[J]. Adv Clin Chem, 2018, 86: 23-70.
[8] Hänzelmann S, Castelo R, Guinney J. GSVA: gene set variation analysis for microarray and RNA-seq data[J]. BMC Bioinformatics, 2013, 14: 7. doi: 10.1186/1471-2105-14-7
[9] Bindea G, Mlecnik B, Tosolini M, et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer[J]. Immunity, 2013, 39(4): 782-795. doi: 10.1016/j.immuni.2013.10.003
[10] Rodríguez-Molinero J, Migueláñez-Medrán BDC, Puente-Gutiérrez C, et al. Association between Oral Cancer and Diet: An Update[J]. Nutrients, 2021, 13(4): 1299. doi: 10.3390/nu13041299
[11] Zhang H, Liu J, Fu X, et al. Identification of Key Genes and Pathways in Tongue Squamous Cell Carcinoma Using Bioinformatics Analysis[J]. Med Sci Monit, 2017, 23: 5924-5932. doi: 10.12659/MSM.905035
[12] Hu Y, Zheng L, Zhang J, et al. LncRNA-MALAT1 is a promising biomarker for prognostic evaluation of tongue squamous cell carcinoma[J]. Eur Arch Otorhinolaryngol, 2020, 277(11): 3155-3160. doi: 10.1007/s00405-020-06023-6
[13] Niu J, Li XM, Wang X, et al. DKK1 inhibits breast cancer cell migration and invasion through suppression of β-catenin/MMP7 signaling pathway[J]. Cancer Cell Int, 2019, 19: 168. doi: 10.1186/s12935-019-0883-1
[14] Hassan NM, Hamada J, Murai T, et al. Aberrant expression of HOX genes in oral dysplasia and squamous cell carcinoma tissues[J]. Oncol Res, 2006, 16(5): 217-224. doi: 10.3727/000000006783981080
[15] Gao H, Li L, Xiao M, et al. Elevated DKK1 expression is an independent unfavorable prognostic indicator of survival in head and neck squamous cell carcinoma[J]. Cancer Manag Res, 2018, 10: 5083-5089. doi: 10.2147/CMAR.S177043
[16] Shi Y, Gong HL, Zhou L, et al. Dickkopf-1 is a novel prognostic biomarker for laryngeal squamous cell carcinoma[J]. Acta Otolaryngol, 2014, 134(7): 753-759. doi: 10.3109/00016489.2014.894251
[17] Qi L, Chen J, Zhou B, et al. HomeoboxC6 promotes metastasis by orchestrating the DKK1/Wnt/β-catenin axis in right-sided colon cancer[J]. Cell Death Dis, 2021, 12(4): 337. doi: 10.1038/s41419-021-03630-x
[18] Zheng W, Qian C, Tang Y, et al. Manipulation of the crosstalk between tumor angiogenesis and immunosuppression in the tumor microenvironment: Insight into the combination therapy of anti-angiogenesis and immune checkpoint blockade[J]. Front Immunol, 2022, 13: 1035323. doi: 10.3389/fimmu.2022.1035323
[19] Liu DX, Wu KH, Zang XY, et al. Dickkopf-related protein 1 as a biomarker of local immune status and worse prognosis of Oral squamous cell carcinoma[J]. Technol Health Care, 2023, 32(3): 1473-1488.
[20] Bonczek O, Krejci P, Izakovicova-Holla L, et al. Tooth agenesis: What do we know and is there a connection to cancer?[J]. Clin Genet, 2021, 99(4): 493-502. doi: 10.1111/cge.13892
[21] Nie Z, Cai S, Wei Z, et al. SH3BGRL2 functions as a crucial tumor suppressor in glioblastoma tumorigenesis[J]. Biochem Biophys Res Commun, 2021, 547: 148-154. doi: 10.1016/j.bbrc.2021.02.035
[22] Dufies M. SH3BGRL2, a new downregulated tumor suppressor in clear cell renal cell carcinomas[J]. EBioMedicine, 2020, 52: 102641. doi: 10.1016/j.ebiom.2020.102641



 下载:
下载: