Expression of Mina53 and CBX8 in Hepatocellular Carcinoma and Their Correlation with Clinicopathological Features and Prognosis
-
摘要:目的
探讨Mina53与CBX8在肝细胞肝癌中的表达,并分析两者与肝细胞肝癌患者临床病理特征及预后的关系。
方法收集121例肝细胞肝癌患者术后切除标本制作组织芯片,采用免疫组织化学PV6000法检测Mina53与CBX8在肝细胞肝癌及相应癌旁相对正常组织中的表达,应用SPSS23.0软件对数据进行统计分析。
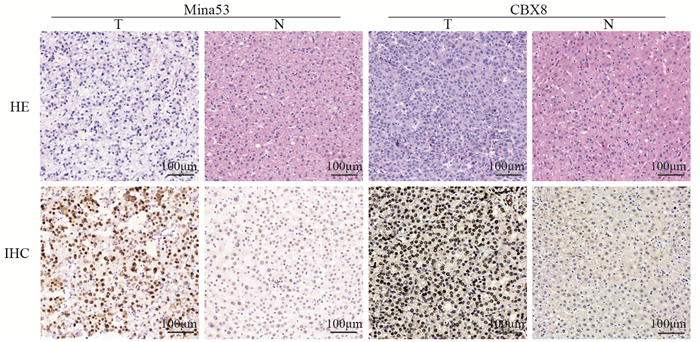
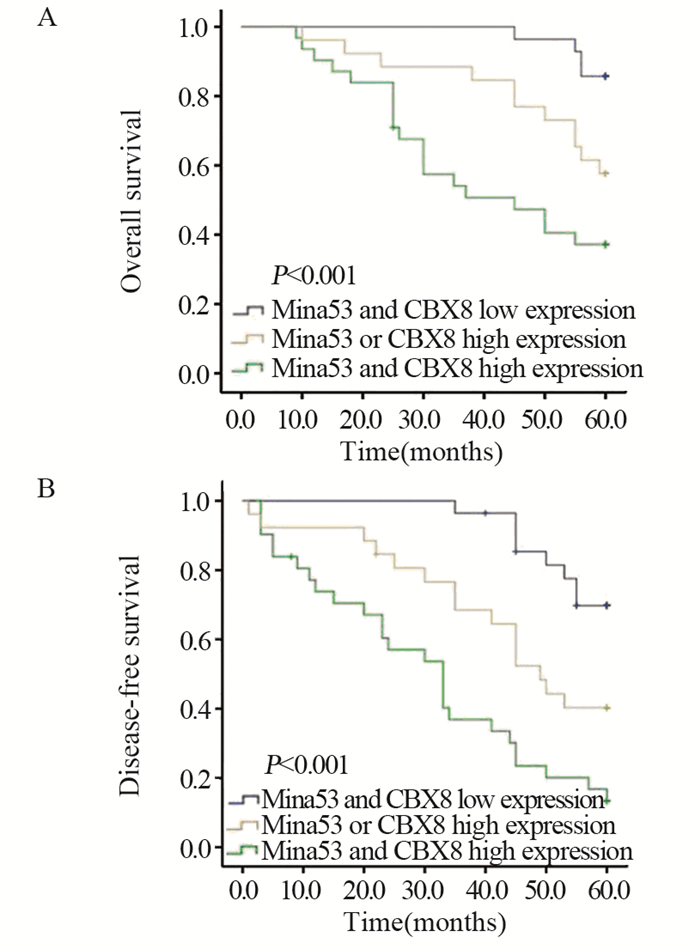
结果Mina53、CBX8在肝细胞肝癌中的表达水平高于癌旁相对正常组织(均P < 0.001)。Mina53表达与肿瘤大小、被膜侵犯、脉管侵犯、病理分级、TNM分期有关(均P < 0.05);CBX8表达与乙肝表面抗原(HBsAg)、肿瘤大小、被膜侵犯、TNM分期有关(均P < 0.05)。肝细胞肝癌中Mina53与CBX8表达呈正相关(r=0.574, P < 0.01)。Mina53与CBX8均高表达患者的OS和DFS较Mina53或CBX8高表达者和两者均低表达者更短(均P < 0.001)。多因素分析显示,Mina53、CBX8的表达水平是肝细胞肝癌患者术后OS和DFS的独立预后影响因素(P < 0.05)。
结论Mina53及CBX8在肝细胞肝癌组织中高表达,可能与肝细胞肝癌的发生发展和浸润转移有关,Mina53和CBX8高表达提示患者预后不良,联合检测Mina53和CBX8有助于肝细胞肝癌的预后判断。
Abstract:ObjectiveTo investigate the expressions of Mina53 and CBX8 in hepatocellular carcinoma (HCC), and to analyze their correlation with the clinicopathological features and prognosis of HCC patients.
MethodsA total of 121 samples of HCC obtained by surgical resection were collected for tissue microarray construction. The expressions of Mina53 and CBX8 were detected by immunohistochemical PV6000 method in HCC and corresponding adjacent non-tumor tissues. Statistical analysis was performed by using SPSS23.0 software.
ResultsThe expressions of Mina53 and CBX8 were both significantly higher in HCC than those in the adjacent non-tumor tissues (both P < 0.001). Expression of Mina53 was correlated with tumor size, capsular invasion, vascular invasion, tumor differentiation and TNM stage (all P < 0.05). Expression of CBX8 was closely correlated with HBsAg, tumor size, capsular invasion and TNM stage (all P < 0.05). The expressions of Mina53 and CBX8 were positively correlated (r=0.574, P < 0.01). Patients with high expressions of both Mina53 and CBX8 had shorter overall survival and disease-free survival than those with high expression of Mina53 or CBX8 or both low expressions of Mina53 and CBX8 (P < 0.05). Multivariate analyses showed that Mina53 and CBX8 expressions were independent influence factors for overall survival and disease-free survival of HCC patients (P < 0.05).
ConclusionThe high expression of Mina53 and CBX8 may be correlated with the pathogenesis, invasion and metastasis of HCC. Patients with high expressions of both Mina53 and CBX8 have a poor prognosis, which indicates that the combined detection of Mina53 and CBX8 is conducive for predicting the prognosis of HCC patients.
-
Key words:
- Mina53 /
- CBX8 /
- HCC /
- Correlation /
- Prognosis
-
0 引言
染色质重构是基因表达动态调控的重要机制之一,主要由不同的蛋白质/蛋白质复合物完成[1]。SWI/SNF复合物是首次在酵母中发现的影响交配型转换的基因复合物之一,转录调控因子等蛋白将SWI/SNF复合物募集到DNA区域,沿DNA移动并通过变构核小体修改DNA的可访问性或可及性(重塑染色质),通过转录调节因子与暴露DNA的结合实现基因表达调控(抑制或者激活)。其亚基基因在20%以上的恶性肿瘤中发生突变[2]。
1 SWI/SNF复合物及其功能
哺乳动物SWI/SNF复合物由多个亚基组成,包括两个催化亚基BRM/SMARCA2和BRG1/SMARCA4,以及一组称为BRG1/BRM相关因子的蛋白质(是SWI/SNF复合物与DNA或蛋白质结合所必需的亚基)[2]。SWI/SNF复合物主要包含两类:BRG1/BRM相关因子复合物(BRG1/BRM associated factor, BAF)及多溴相关BAF复合物(polybromo-associated BAF, PBAF)。BAF及PBAF均包括三个核心亚基(SMARCB1、SMARCC1及SMARCC 2),辅助调节亚基存在差异,BAF主要包括ARID1A/1B、DPF1/2/3、SS18、SMARCE1、SMARCD1/D2/D3、ACTL6A及BRD9,PBAF主要包括ARID2、PBRM1、PHF10、SMARCE1、SMARCD1/D2/D3、ACTL6A及BRD7。SMARCA2及SMARCA4含有6个保守结构域:QLQ结构域、富含脯氨酸的结构域、小解旋酶/SANT相关结构域、DNA依赖性ATP酶结构域、视网膜母细胞瘤(RB)结合结构域(LxCxE)和Bromo结构域。Bromo结构域与乙酰化组蛋白相互作用,参与SWI/SNF复合物与DNA的结合和稳定性。LxCxE结构域与RB肿瘤抑制基因家族的成员结合,而QLQ结构域与蛋白质-蛋白质相互作用有关。最后,解旋酶和DExDc结构域分离DNA双链,需ATP水解[3]。但这两种ATP水解酶在功能上存在显著差别,并不能彼此弥补。INI 1/SMARCB 1、BAF 155/SMARCC1和BAF170/SMARCC2称为“核心亚基”,其对于SMARCA2或SMARCA4的ATP依赖染色质重塑活性是必需的,主要参与双链断裂和核苷酸切除修复。SWI/SNF复合物还有7~10个辅助调节亚基,靶向特定的DNA或基因位点,负责不同复合物靶向的特异性基因组。BAF复合物包含ARID1A/B,PBAF复合物包含PBRM1、ARID2、BRD7和PHF10[4]。其含有与DNA或组蛋白相互作用所需的特定结构域(溴结构域、染色质结构域、DNA结合结构域、ARID及Zing finger等)[2-4]。
SWI/SNF亚基不仅与启动子结合,而且与其他调控区域(如增强子和DNA复制起始区)紧密结合。此外,SWI/SNF可结合/共沉淀许多蛋白质,在细胞周期、细胞骨架和染色体组织等过程中发挥作用[3]。表明SWI/SNF复合物的功能比单纯的转录调控更为广泛。
2 SWI/SNF复合物亚基变异在肿瘤中的作用
现针对各个SWI/SNF复合物亚基变异进行分析,见表 1[3, 5]。
表 1 SWI/SNF复合物亚基在恶性肿瘤中的变异情况Table 1 Abnormality of SWI/SNF complex subunits in malignant tumors
2.1 催化亚基
SMARCA2和SMARCA4虽同为SWI/SNF的ATP水解酶,但两者在恶性肿瘤中的突变谱明显不同。SMARCA4和SMARCA2在结直肠癌、浆液性卵巢癌、胰腺癌、肝细胞癌、肺癌和乳腺癌、胶质瘤、黑色素瘤和鳞状细胞癌中均有不同频率的突变,在卵巢透明细胞癌、肾癌、血液系统肿瘤和髓母细胞瘤中存在SMARCA4突变但无SMARCA2突变[5-6],大多数突变位于解旋酶催化结构域。SMARCA4是继ARID1A之后恶性肿瘤中第二频繁突变的SWI/SNF基因,其作用比SMARCA2更重要。
在卵巢高钙血症型小细胞癌(small cell carcinoma of the ovary, SCCOHT)中均有SMARCA2蛋白缺失[7],常发生表观基因改变。SMARCA2缺失可发生在横纹肌样瘤、乳腺癌、肺癌、食道癌、膀胱癌、胃癌和卵巢癌中[7-8]。SMARCA2突变非常罕见,但可见于腺样囊性癌(5%)[9]。SMARCA4和SMARCA2是SWI/SNF的相对互斥亚基,在SMARCA4缺陷肿瘤中,SMARCA2上调,可补充SMARCA4的功能丢失[4]。但在SCCOHT和SMARCA4缺陷性胸部肉瘤(SMARCA4-deficient thoracic sarcomas, SMARCA4-DTS)中SMARCA4和SMARCA2表达同时丢失[10],与在其他SMARCA4缺陷肿瘤中的合成致死关系不同。
在SCCOHT中均有SMARCA4基因突变。SCCOHT是双等位SMARCA4失活突变(包括截短、移码或缺失),近一半的SCOOHT在种系细胞中检测出SMARCA4突变,并可能表现为横纹肌样瘤易感性综合征[11]。SMARCA4-DTS也与重复性SMARCA4突变相关,导致SMARCA4功能丧失[12]。
催化亚基突变也出现在胃肠道低分化癌(undifferentiated gastrointestinal carcinoma, UGC)中,包括:结肠、小肠、胃和远端食道,主要显示SMARCB1或SMARCA4失活,也有SMARCA2和ARID1A改变。SMARCA2和SMARCB1是UGC中最常见的SWI/SNF突变亚基(分别为77%和50%)。所有SMARCB1突变的病例也有SMARCA2突变,而SMARCA2和SMARCA4突变相互排斥,SMARCB1和SMARCA4之间也是如此[3]。
2.2 核心亚基
SMARCB1几乎在所有恶性横纹肌样肿瘤(malignant rhabdoid tumor, MRT)中都存在基因缺失或截短突变,是MRT中唯一的重复性遗传异常[13]。在家族性神经瘤病和脑膜瘤中有SMARCB1双等位基因丢失。80%以上的上皮样肿瘤中也存在SMARCB1蛋白丢失(主要因纯合子缺失)。而其他两个核心亚基(SMARCC1和SMARCC2)却鲜有突变[5]。
在MRT中,SMARCB1缺失增加了多梳抑制复合物2(polycomb repressive complex 2, PRC2)亚基EZH2的丰度,EZH2高表达使PRC2不能被置换,导致DNA甲基化,从而抑制肿瘤抑制基因CDKN2A,导致增殖增加[14]。
在肾髓质癌[15]中存在不平衡易位后的SMARCB1截短。SMARCB1功能丧失可诱导细胞周期蛋白D1,导致细胞周期的G1期细胞增殖。SMARCB1缺陷约占鼻窦癌的10%[16]。与MRT一样,其基因组高度稳定,表明SMARCB1丢失可能是其发生的驱动因素。低分化小儿脊索瘤存在由染色体22q11缺失引起的SMARCB1缺失[17]。
2.3 辅助调节亚基
在家族性脊膜瘤中,SMARCE1杂合突变并完全丢失,并与透明细胞组织学形态相关[18],更具侵袭性,较易扩散转移,可能与SMARCE1作用于类固醇激素反应有关[19]。在透明细胞肾癌(约40%)、上皮样肉瘤(约83%)和胆管癌(约32%)中存在PBRM1失活突变或缺失。14%的HCV相关肝细胞肝癌、小部分恶性黑色素细胞瘤和肺癌有ARID2失活突变[5]。
ARID1A是肿瘤中突变频率最高的SWI/SNF基因,一项约3 000份肿瘤标本的大型研究发现,其在10%以上的肿瘤中存在丢失:胃癌(14%)、间变性甲状腺癌(14%)、低级别和高级别子宫内膜样癌(分别占29%和39%)、子宫癌(浆液癌18%、癌肉瘤14%)[5]。并在肝细胞癌[20]、肺腺癌[21]、膀胱癌[22]或胆管癌[23]中存在突变。约50%的卵巢透明细胞癌及近40%的子宫内膜癌存在ARID1A突变,其也与良性子宫内膜异位症癌变相关[24]。ARID1A在肿瘤中常发生截短突变,导致C末端结合区的缺失,破坏复合物的组合。ARID1A丢失并不能通过ARID1B的表达增多加以补偿,且在大多数肿瘤中ARID1B表达比ARID1A更低[25]。
SMARCC2突变很罕见,但可见于微卫星不稳定的胃癌和结直肠癌[26]。SKARCC2外显子8的重复序列是移码突变的热点,分别存在于胃癌(9%)和结直肠癌(15%)中。该突变产生终止密码子,导致SMARCC2的功能丧失。
2.4 滑膜肉瘤SS18移位
SS18首先在滑膜肉瘤(synovial sarcoma, SS)中被鉴定,t(X; 18)(p11.2; q11.2)易位引起18号染色体上的SS18与X染色体上的SSX1、SSX2或SSX4融合[27],导致SS18 C末端与SSX蛋白的78个C末端氨基酸融合。SS18-SSX融合蛋白与正常SS18亚基竞争结合到SWI/SNF复合物中。可能由于融合蛋白体积较大,SMARCB1亚基从滑膜肉瘤BAF(ssBAF)复合物中被置换。通常,SMARCB1被排出及降解导致功能丧失,然而,SW1/SNF复合物中的SS18-SSX进入整合导致复合物功能增强,即染色质占据能力增强和PRC被强力驱逐置换。融合蛋白的SSX与特定DNA位点结合可导致ssBAF定向到其他的基因组位点[28],激活SOX2致癌基因。该机制使SWI/SNF复合物具有了致癌活性。
3 其他SWI/SNF相关肿瘤发生机制
SWI/SNF复合物可通过与长链非编码RNA(lncRNA)相互作用参与肿瘤发生。其机制是:lncRNA直接与SWI/SNF复合物相互作用,拮抗其活性,或使SWI/SNF复合物在某些特定位点募集[29],促使肿瘤发生。
最近研究成果包括:(1)发现SWI/SNF复合物在谱系特异性基因增强子调节中的作用[30]。SWI/SNF复合物直接与p300组蛋白乙酰转移酶结合,从而调节靶基因上H3K27Ac(组蛋白H3K27乙酰化)的活性。这种SWI/SNF调节在典型的远端增强子中显示出强活性,主要参与分化和发育的基因。提示SWI/SNF亚基缺失(如SMARCB1、ARID1A)可通过该途径诱导肿瘤发生; (2)如前所述,SWI/SNF复合物(如SMARCB1)与PRC2的EZH2之间存在特异性拮抗关系,多梳复合物是SWI/SNF的关键非核小体底物。EZH2负责组蛋白H3第27位赖氨酸的三甲基化,这是与基因组转录抑制相关的标记[14]。
4 SWI/SNF复合物亚基驱动相关肿瘤的新型治疗策略探讨
在临床上,对异常缺失的基因产物进行恢复很难实现,因此,对于基因表达缺失驱动肿瘤的治疗仍然存在很大的挑战。但SWI/SNF亚基异常与其他致癌基因异常共同发生,因此,利用特异性的合成致死关系是其治疗的有效途径。合成致死关系是导致细胞适应性丧失的两种遗传事件(如突变)的组合,即两者同时发生将会导致细胞死亡。目前,对于SWI/SNF复合物亚基之间或SWI/SNF亚基缺失与异常激活的致癌信号途径之间的合成致死关系已经成为精准治疗的热门领域。目前主要如下。
4.1 SWI/SNF复合物旁系同源亚基内部之间
SWI/SNF亚基缺失肿瘤会表现出对其他旁系同源亚基独特的强依赖性,以维持其增殖适应性。如SMARCA4-SMARCA2及ARID1A-ARID1B。在SMARCA4缺陷非小细胞肺癌(NSCLC)细胞系中,RNAi文库合成致死筛选发现,SMARCA2为癌细胞系增殖所需的最强合成致死依赖性亚基[31]。可使用靶生物分子的配体依赖性蛋白质降解技术特异性降解亚基,直接靶向SWI/SNF ATP酶和溴结构域[32]。
4.2 SMARCB1表达缺失和EZH2抑制
SMARCB1表达缺失和PRC2抑制之间存在合成致死关系,EZH2是PRC2的催化亚基。小分子介导EZH2抑制的临床前研究已在SMARCB1缺陷肿瘤中得到验证[33]。而且,特异性EZH2抑制小分子还可与其他关键效应信号抑制剂组合使用,以提高效果,如溴结构域或SHH信号抑制剂[32]。
4.3 SWI/SNF亚基缺失与其他下游依赖性信号通路
在SWI/SNF亚基缺失肿瘤中,发现了多个其他下游依赖性信号通路的合成致死关系靶标,如YES1[34]、PARP [35]、ATR[36]、PIK3CA[37]、KRAS[38]、BRAF[3]、Topo Ⅱ[39]及细胞周期蛋白D1[40]等。
SWI/SNF复合物与DNA损伤修复有关。SWI/SNF亚基缺失癌细胞对DNA修复途径抑制剂的易感性增强。PIK3/AKT途径在该领域具有重要意义。基于shRNA的筛选研究发现,PIK3CA(p110)和PIK3R2(p85)的两个催化亚基在ARID1A缺陷癌细胞中显示很高的脆弱性。抑制PIK3途径药物可能是易实现的临床选择。事实上,一些针对PIK3/AKT途径节点的制剂已快速转向FDA批准[41]。SMARCA4表达缺失与对拓扑异构酶Ⅱ抑制剂的耐受性相关,EZH2抑制剂与依托泊苷(一种拓扑异构酶Ⅱ(Topo Ⅱ)抑制剂)在SMARCA4缺失肿瘤中具有协同抗肿瘤作用[42]。SMARCB1失活可导致细胞周期蛋白D1上调。目前一种细胞周期蛋白依赖性激酶(CDK)4/6抑制剂ribociclib(LEE011)正在进行阶段1/2临床试验[40]。
4.4 其他
目前,针对滑膜肉瘤中致癌性SS18-SSX特异性降解或针对SS18-SSX的合成致死关系的治疗途径还在研究中。此外,SWI/SNF亚基还可作为治疗反应的预测因子。如类固醇治疗对小儿急性淋巴细胞白血病的反应与3个SWI/SNF亚基(SMARCB1、SMARCA4、ARID1A)的表达水平相关:低表达与较高的治疗反应相关[43]。SMARCE1表达可用作卵巢癌和肺癌药物反应的标志物:卵巢癌对顺铂、多柔比星和5-氟尿嘧啶的敏感度可能与低SMARCE1表达有关[44],低SMARCE1表达也与非小细胞肺癌的MET和ALK抑制剂耐药相关[45]。
5 总结
SWI/SNF复合物在细胞信号调控等多个方面发挥了重要作用,并且,其亚基异常涉及较多恶性肿瘤的发生发展,因此,对其复杂的作用机制及临床应用开发研究很有意义,如进一步研究SWI/SNF复合物异常对恶性肿瘤预后的影响、其表达缺失的具体机制(如SNP、插入缺失、拷贝数变异、体细胞DNA突变、甲基化/染色质结构/基因表达水平)及致癌机制(包括单个及多个亚基),基因组学时代的来临也使系统研究SWI/SNF复合物异常涉及的信号通路、合成致死筛选及具体机制成为可能,从而为此类肿瘤的靶向精准治疗带来希望。
作者贡献叶冬雪:实验实施,采集、分析数据,起草文章刘芬:实验实施,分析数据杨茜:采集数据付海洋:实验实施周璇:获取研究经费,设计指导实验项锋钢:设计实验,指导、修改论文 -
表 1 Mina53、CBX8在肝细胞肝癌和相应癌旁相对正常组织中表达的比较
Table 1 Comparison of Mina53 and CBX8 expressions in HCC and adjacent non-tumor tissues

表 2 Mina53、CBX8的表达与肝细胞肝癌患者临床病理特征的关系
Table 2 Correlation of Mina53 and CBX8 expressions with clinicopathological features of HCC patients

表 3 肝细胞肝癌组织中Mina53与CBX8表达的相关性
Table 3 Correlation between expressions of Mina53 and CBX8 in HCC tissues

表 4 单因素和多因素Cox回归模型分析影响肝细胞肝癌患者总体生存时间的因素
Table 4 Univariate and multivariate Cox regression analyses of influence factors for overall survival of HCC patients

表 5 单因素和多因素Cox回归模型分析影响肝细胞肝癌患者无疾病生存时间的因素
Table 5 Univariate and multivariate Cox regression analyses of influence factors for disease-free survival of HCC patients

-
[1] Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7-30. doi: 10.3322/caac.21442
[2] Lim C, Dejong CH, Farges O. Improving the quality of liver resection: a systematic review and critical analysis of the available prognostic models[J]. HPB (Oxford), 2015, 17(3): 209-221. doi: 10.1111/hpb.12346
[3] Hartke J, Johnson M, Ghabril M. The diagnosis and treatment of hepatocellular carcinoma[J]. Semin Diagn Pathol, 2017, 34(2): 153-159. doi: 10.1053/j.semdp.2016.12.011
[4] Di Croce L, Helin K. Transcriptional regulation by Polycomb group proteins[J]. Nat Struct Mol Biol, 2013, 20(10): 1147-1155. doi: 10.1038/nsmb.2669
[5] Song X, Tang T, Li C, et al. CBX8 and CD96 Are Important Prognostic Biomarkers of Colorectal Cancer[J]. Med Sci Monit, 2018, 24: 7820-7827. doi: 10.12659/MSM.908656
[6] Yuan GJ, Chen X, Lu J, et al. Chromobox homolog 8 is a predictor of muscle invasive bladder cancer and promotes cell proliferation by repressing the p53 pathway[J]. Cancer Sci, 2017, 108(11): 2166-2175. doi: 10.1111/cas.13383
[7] Zhang L, Zhou Y, Cheng C, et al. Genomic analyses reveal mutational signatures and frequently altered genes in esophageal squamous cell carcinoma[J]. Am J Hum Genet, 2015, 96(4): 597-611. doi: 10.1016/j.ajhg.2015.02.017
[8] Tang B, Tian Y, Liao Y, et al. CBX8 exhibits oncogenic properties and serves as a prognostic factor in hepatocellular carcinoma[J]. Cell Death Dis, 2019, 10(2): 52. doi: 10.1038/s41419-018-1288-0
[9] Makoto T, Yoshiro K, Mikiko S, et al. A novel myc target gene, mina53, that is involved in cell proliferation[J]. J Biol Chem, 2002, 277(38): 35450-35459. doi: 10.1074/jbc.M204458200
[10] Chitra T, Fei C. Current understanding of mdig/MINA in human cancers[J]. Genes Cancer, 2015, 6(7-8): 288-302. http://cn.bing.com/academic/profile?id=9a4025243cbad88c33e7a8ae3ec84af1&encoded=0&v=paper_preview&mkt=zh-cn
[11] Fujino S, Kinugasa T, Sudo T, et al. Mina53 nuclear localization is an important indicator of prognosis in patients with colorectal cancer after adjuvant chemotherapy[J]. Oncol Rep, 2018, 40(1): 101-110. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=e5a47d09ea0e47d6802c82556134b28e
[12] Ferreira MJ, Pires-Luís AS, Vieira-Coimbra M, et al. SETDB2 and RIOX2 are differentially expressed among renal cell tumor subtypes, associating with prognosis and metastization[J]. Epigenetics, 2017, 12(12): 1057-1064. doi: 10.1080/15592294.2017.1385685
[13] Kumar SA, Thakur C, Li L, et al. Pathological and prognostic role of mdig in pancreatic cancer[J]. Genes Cancer, 2017, 8(7-8): 650-658. http://europepmc.org/abstract/MED/28966726
[14] Thakur C, Lu YJ, Sun JY, et al. Increased expression of mdig predicts poorer survival of the breast cancer patients[J]. Gene, 2014, 535(2): 218-224. doi: 10.1016/j.gene.2013.11.031
[15] Zhou LX, Zhang T, Zhu Q, et al. Mina53 regulates the differentiation and proliferation of leukemia cells[J]. Hematol Oncol, 2019, 37(4):513-515. doi: 10.1002/hon.2621
[16] Luo W, Zhu XN, Liu W, et al. MYC associated zinc finger protein promotes the invasion and metastasis of hepatocellular carcinoma by inducing epithelial mesenchymal transition[J]. Oncotarget, 2016, 7(52): 86420-86432. doi: 10.18632/oncotarget.13416
[17] Accari SL, Fisher PR. Emerging Roles of JmjC Domain-Containing Proteins[J]. Int Rev Cell Mol Biol, 2015, 319: 165-220. doi: 10.1016/bs.ircmb.2015.07.003
[18] Cao PL, Kumagai N, Inoue T, et al. JmjC Domain-Encoding Genes Are Conserved in Highly Regenerative Metazoans and Are Associated with Planarian Whole-Body Regeneration[J]. Genome Biol Evol, 2019, 11(2): 552-564. doi: 10.1093/gbe/evz021
[19] Ogasawara S, Komuta M, Nakashima O, et al. Accelerated expression of a Myc target gene Mina53 in aggressive hepatocellular carcinoma[J]. Hepatol Res, 2010, 40(3): 330-336. doi: 10.1111/j.1872-034X.2009.00604.x
[20] Zhang CZ, Chen SL, Wang CH, et al. CBX8 Exhibits Oncogenic Activity via AKT/beta-Catenin Activation in Hepatocellular Carcinoma[J]. Cancer Res, 2018, 78(1): 51-63. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=088627128fab7e8e5454a57e75773681
[21] Xiao LS, Zhou ZX, Li WW, et al. Chromobox homolog 8 (CBX8) Interacts with Y-Box binding protein 1 (YBX1) to promote cellular proliferation in hepatocellular carcinoma cells[J]. Aging (Albany NY), 2019, 11(17): 7123-7149. http://cn.bing.com/academic/profile?id=7ae4f5e9c61e606e84705c318f9ec26f&encoded=0&v=paper_preview&mkt=zh-cn
[22] Wang W, Lu Y, Stemmer PM, et al. The proteomic investigation reveals interaction of mdig protein with the machinery of DNA double-strand break repair[J]. Oncotarget, 2015, 6(29): 28269-28281. doi: 10.18632/oncotarget.4961
[23] Yang SB, Liu WH, Li MY, et al. Insulin-Like Growth Factor-1 Modulates Polycomb Cbx8 Expression and Inhibits Colon Cancer Cell Apoptosis[J]. Cell Biochem Biophys, 2015, 71(3): 1503-1507. doi: 10.1007/s12013-014-0373-y



 下载:
下载: